A evolução do receptor TAS2R38 em primatas: O amargor da seleção natural

O amargor dos alimentos pode ser uma pista de sua toxicidade, por isso receptores proteicos que conseguem detectar moléculas associadas ao gosto amargo, como os TAS2Rs permitem aos animais que os expressam evitarem toxinas em seu meio ambiente, principalmente compostos químicos nocivos de defesa produzidos pelas plantas. Desta maneira, receptores como TAS2Rs estariam sob complexas pressões de seleção natural.
Stephen Wooding investigou algumas dessas pressões, ao examinar as assinaturas moleculares dessas pressões que estariam agindo sobre os fenótipos especificados pelo gene TAS2R38 em diversas espécies de primatas, através do sequenciamento completo do gene (1.002 pares de bases) de 40 espécies, representando todos os grandes táxons de primatas. Seus resultados revelaram extensa variação nesse locus, com substituições de um nucleotídeo por outro ocorrendo em 448 posições diferentes, sendo que destas 201 resultaram em alterações dos aminoácidos codificados, as chamadas substituições não-sinônimas. Além disso, Wooding identificou duas deleções de nucleotídeos únicos, uma deleção de três nucleotídeos ao mesmo tempo preservando o marco de leitura, e uma resultando em um códon de parada prematuro.
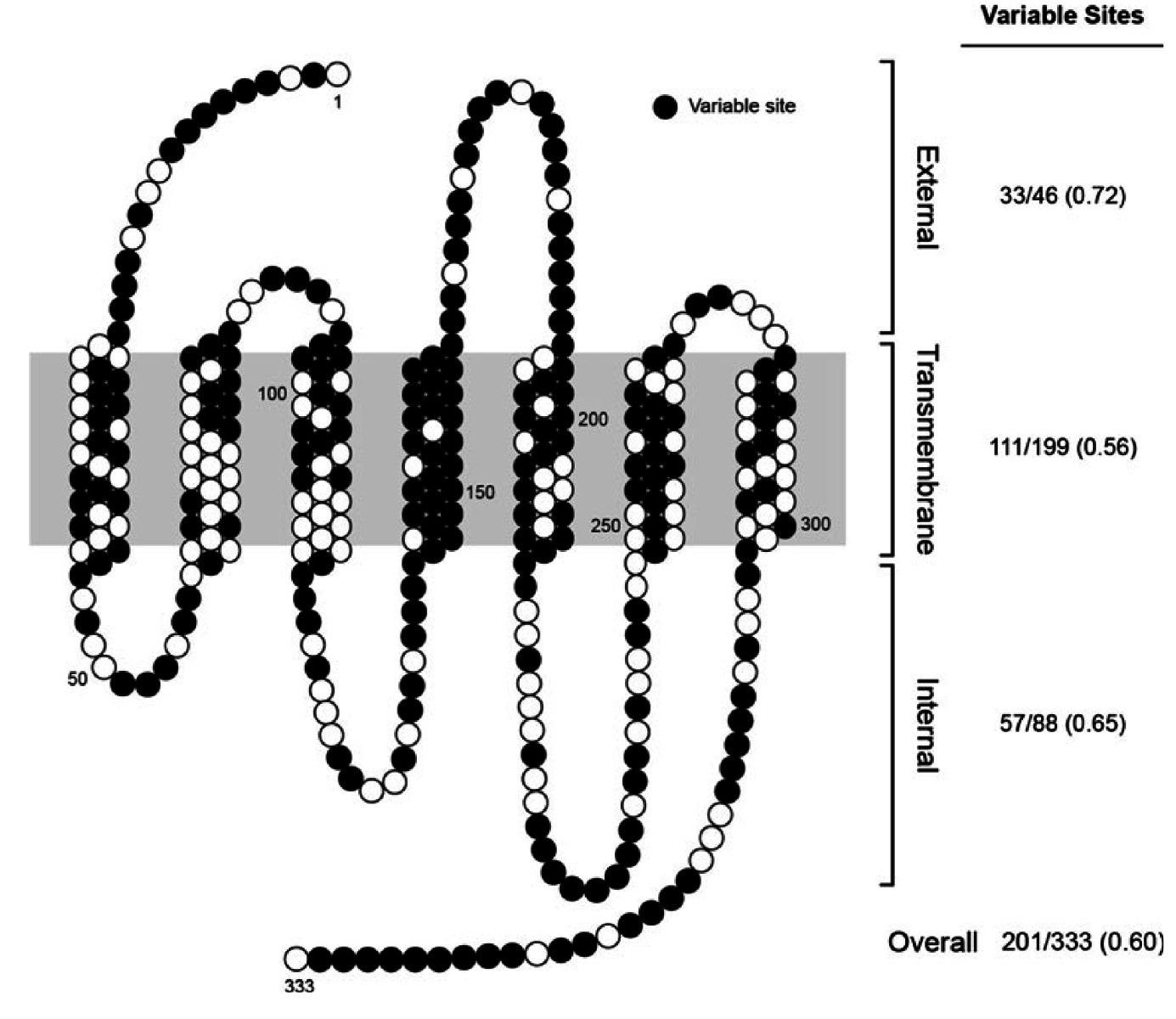
Ao analisar as 37 espécies de primatas (as análises excluíram três espécies com códons de parada alterados, que são inadequados para modelagem ω) das 40 originais que possuíam genes TAS2R38 intactas foi verificado que das 1,002 posições, correspondendo ao número de bases que formam o gene, 448 delas variava (45%) resultando em 201 alterações das 333 posições de aminoácidos (60%). E apesar de sítios variáveis se distribuírem através de toda a extensão do gene, havia proporcionalmente mais substituições nas posições que codificam os aminoácidos das alças externas que variavam em 72% das posições (isto é, em 33 dos 46 resíduos). Em contraste, poucas substituições forma identificadas em regiões correspondentes aos domínios transmembrana que variaram, em 56% dos casos (i.e., 111 dos 199 resíduos), alças internas que variavam em 65% das posições, ou seja (ou seja, em 57 dos 88 resíduos).
A figura abaixo mostra as posições variáveis dos resíduos de aminoácidos nas 37 espécies analisadas utilizando-se o software PAML. Os círculos indicam a posição relativa dos resíduos de aminoácidos no receptor de TAS2R38 a partir do resíduo N-terminal (posição 1) em direção ao C-terminal (posição 333). Os sítios transmembrana, externos e internos foram categorizados seguindo Floriano et al. (2006; citado por Wooding). Já os círculos preenchidos mostram se a variação estava presente na posição determinada no alinhamento global das sequências de primatas, isto é, se mais de um tipo de aminoácido foi observado naquela posição específica. Para cada categoria de sítio (transmembrana, externo e interno), os valores à direita indicam o número de sítios na categoria (denominador), o número de sítios variáveis (numerador), e a fracção de sítios variáveis (entre parênteses).
Como forma de identificar as assinaturas da seleção natural foram analisadas as taxas relativas de substituições sinônimas (i.e. que não envolvem a mudança do aminoácido codificado) e não-sinônimas (i.e. que envolvem a mudança do aminoácido codificado), ω (= dN/dS), que refletem as pressões seletivas de longo prazo as quais estão sujeitos os organismos em função dos fenótipos a eles conferidos por seus genes, o que acaba deixa marcas nos padrões de abundância relativa de certos tipos de substituições.
O ponto chave é que sob neutralidade - isto é, caso as substituições estivessem mudando de forma a não impactar funcionalmente significativamente as biomoléculas codificadas e, por isso, não interferindo, de maneira apreciável, nas diferenças no sucesso reprodutivo dos portadores de uma ou de outra variante - se esperaria que as taxas de variação sinônima e não-sinônima fossem bem semelhantes, aproximadamente iguais, de tal modo que ω = 1.
Por outro lado, caso estivesse ocorrendo o que alguns chamam de seleção purificadora - isto é, as mudanças que alterassem os resíduos de aminoácidos codificados estivessem causando alterações na função e estrutura das proteínas de modo que seus portadores estivessem em desvantagem em termos de sobrevivência e reprodução - esperaríamos que as variantes não-sinônimas fossem purgadas das populações, diminuindo assim ω (<1). Em contraste, caso estivesse ocorrendo a seleção positiva, seriam as variantes não-sinônimas as favorecidas, já que as alterações em resíduos de certos aminoácidos estariam tendo um impacto positivo na sobrevivência e sucesso reprodutivo dos indivíduos que as portassem, ao alterar a estrutura e função das proteínas codificadas pelo gene em questão, aumentando, assim, ω (>1). Para maiores detalhes sobre os testes de identificação de marcas moleculares da seleção usando modelos de evolução neutros, indico o artigo do evolucionismo, "Marcas da adaptação: A teoria neutra e as assinaturas moleculares da seleção natural" em que mais detalhes são dados sobre este tipo de estratégia de análise evolutiva.
Quando Wooding analisou a assinatura da seleção natural no gene TAS2R38 como um todo, encontrou ω = 0,60, um valor elevado quando comparado ao de outros genes, mas significativamente menor do que seria esperado caso estas substituições estivessem ocorrendo de maneira neutra (valor p = 4,0x10-9), indicando que a seleção purificadora estaria mantendo a estrutura básica do receptor ao eliminar os indivíduos cujas substituições provocassem alterações funcionais relevantes. Mas como enfatiza Wooding, essas tendências podem ser localizadas, afetando apenas de maneira distinta regiões diferentes dos genes.
Por exemplo, ao estimar ω para o gene DRB de mamíferos do MHC da classe II que participa na resposta imune, encontram-se valores consistentes com uma pressão global de seleção purificadora. Porém, regiões envolvidas no reconhecimento de antígenos mostram padrões de substituições que constituem-se em assinaturas de seleção positiva, algo similar ao encontrado em um estudo de divergência entre os loci TAS2R em seres humanos. Por causa disso, Wooding executou testes para estes tipos de efeitos localizados no gene TAS2R38, concentrando-se em regiões que codificam as principais categorias funcionais e pontos chave para estrutura do receptor. Neste caso, por exemplo, as taxas de substituições não-sinônimas encontradas foram significativamente menores do que o esperado, em domínios transmembrana ( ω = 0,55, valor p = 1.18 × 10-12) e em alças internas (ω = 0,51, valor p = 7,04 × 10-5), mas não nas alças externas (ω = 1,16 , valor p = 0,53), sendo que evidências robustas de seleção positiva foram encontradas para a alça externa 2 da proteína (ω = 2,53, valor p = 0,02), valor consistente com mudanças rápidas nos padrões de ligação da molécula alvo do receptor.
A figura ao lado exibe alguns testes para a neutralidade dirigidos a ‘marcos’ funcionais e estruturais específicos. Em (a) podemos observar uma estrutura secundária e os marcos funcionais previstos para o receptor TAS2R38 proposto por Floriano et al. (2006) (citado por Wooding); e em (b) as verossimilhanças, estimativas de parâmetros e valores p obtidos para cada estrutura independentemente sob o Modelo de 0 (ω = 1) e o Modelo de 0 (um ω).
Os autores defendem que estes resultados sugerem que ocorreram rápidas mudanças, ainda que restringidas por questões estruturais e funcionais, ao longo da história evolutiva das respostas ao sabor amargo durante da evolução dos primatas. Isso mostra como somos cada vez mais capazes de olhar para os detlahes do processo de evolução molecular e destrinchar seus diversos componentes, sejam eles neutros, adaptativos ou funcionalmente conservativos, nos dando uma melhor perspectiva de como se dá a evolução biológica ao correlacioná-los com os fenótipos dos organimos.
______________________________________________________
Referências:
Wooding, S. (2012). Signatures of Natural Selection in a Primate Bitter Taste Receptor Journal of Molecular Evolution, 73 (5-6), 257-265 DOI: 10.1007/s00239-011-9481-0
