É a evolução genética previsível? Parte I

As ciências modernas são empreitadas intelectuais e sociais bastante complexas, dificilmente contidas pela camisa de força de um “método científico” único e estanque. Os desenvolvimentos históricos, o contexto tecnológico e a criatividade, são ingredientes importantíssimos que também fazem parte desta federação de formas de investigação (para usar a expressão de Susan Haack) a qual denominamos “ciência”.

Esta heterogeneidade não impede que reconheçamos as diversas disciplinas científicas e as diferenciemos de outras empreitadas e atividades intelectuais humanas. Os cientistas lançam mão de vários métodos específicos de investigação, que constituem-se em uma série de ferramentas heurísticas que vão desde estratégias de inferência variadas (indução, dedução, abdução), testes estatísticos, modelos matemáticos, simulações computacionais, protocolos para estudos observacionais e experimentais controlados, métodos analíticos e comparativos, esquemas causais de modelagem etc. Mas além disso, as ciências valem-se de ferramentas cognitivas de avaliação e escolha de teorias e hipóteses, que podem ser vistas como 'virtudes cognitivas' que se mostraram eficientes na condução da pesquisa científica. Entre estas é possível citar várias como: poder explanatório, precisão, reprodutibilidade, escopo e abrangência, coerência inter-teórica, fecundidade, etc além da principal virtude epistêmica, a acessibilidade empírica, especialmente via testes controlados que dependem da implementação dos métodos antes mencionados.
As teorias físicas mais tradicionais, estruturadas de forma mais rigorosa e baseadas em leis matemáticas e probabilísticas muito precisas (amplamente válidas e testáveis em condições muito controladas e idealizadas), obtém muito de seu status de seu poder preditivo, como nos casos da relatividade geral e da mecânica quântica, e mesmo como (foi) é o caso da mecânica newtoniana. Nestas áreas o modelo nomológico-dedutivo tem sua melhor representação de instanciamento. Porém a ciência é mais do que isso.
Diferentemente das teorias físicas tradicionais as teorias biológicas sobre evolução concentram-se em seu conteúdo explanatório e na possibilidade de 'retrodizer' fenômenos, ao invés de predizê-los, como as ciências físicas (e mesmo a biologia experimental) fazem, o que é óbvio pois a evolução é uma ciência histórica, como a arqueologia, a geologia, bem como parte da astronomia planetária e, até mesmo, da cosmologia, guardando as devidas diferenças em termos de precisão e nível de matematização (veja os artigos de Carol C. Cleland sobre os assunto nas referências).
As explicações de eventos passados integram os resultados experimentais e analíticos (de diversas outras áreas, tais como biologia molecular e bioquímica, fisiologia, anatomia e fisiologia comparativa funcional, biologia do desenvolvimento, sistemática, ecologia, biogeografia, paleobiologia etc) em um quadro mecanicístico mais geral que reúne narrativas históricas em um arcabouço estatístico elaborado em termos da genética de populações e da genética quantitativa. Estas diferentes áreas e evidências são amarradas em um contexto abdutivo, que permite testes empíricos, formulação de hipóteses adicionais e investigação analítica mais aprofundada, através do que se convencionou chamar de 'Inferência pela melhor explicação'.
Futurologia, ou seja extrapolação de 'tendências' históricas para o futuro, em ciências históricas ainda é algo, no mínimo, bastante controverso. As inúmeras cadeias causas que se encontram nos processos históricos, sejam eles a história social, política ou cultural humana, a geologia, a paleobiologia, a origem de sistemas planetários ou a evolução da vida, tornam estes processos tremendamente contingentes e, portanto, muito difíceis de prever. Para muitos estas considerações impõem limites severos a disciplinas como a biologia evolutiva.
Esta situação, entretanto, está longe de ser um consenso inabalável dentro da própria comunidade científica e filosófica, por isso talvez não seja algo que precisemos nos conformar. Estudos em evolução experimental tanto em campo como em laboratório (especialmente usando populações de microorganismo como os efetuados pelo grupo de Richard E. Lenski na Michigan State University) e as plataformas de AL (Artificial Life), como Avida e Polyword sugerem uma tendência diferente. Por isso, mesmo com a consciência de que a aleatoriedade, juntamente com outros processos estocásticos e contingentes, tem importância inegável durante a evolução biológica, alguns fenômenos indicam que a evolução biológica pode ter alguns aspectos replicáveis e até predizíveis.
A evolução convergente e paralela são os principais exemplos deste tipo de fenômeno e, desde muito tempo, levaram os cientistas a especularem sobre 'leis gerais' da evolução. Alguns padrões fósseis expressos na forma em que certas linhagens se ramificam e se extinguem também sugerem que características associadas as estratégias reprodutivas (K-selecionistas vs r-selecionistas) e de forrageio (generalistas vs especialistas) também possam indicar padrões gerais reprodutíveis, talvez mesmo definidos por processos de níveis hierárquicos mais elevados, como a 'seleção de espécies' ou de clados.
Ao mesmo tempo, exemplos de particularidades e singularidades históricas na evolução são também muito conhecidos, especialmente os padrões de sobrevivência relacionados às grandes extinções. Stephen Jay Gould batizou um de seus livros mais interessantes de 'Wonderful Life' ('Vida Maravilhosa'), inspirado no filme It's a Wonderful Life (em português "A felicidade não se compra") de Frank Capra, estrelado por James Stewart. No filme George Bailey, o personagem de James Stewart, um frustrado e desesperado homem de negócios, recebe ajuda de um anjo que mostra a ele como seria a vida das pessoas, com que ele teve contato, caso ele jamais tivesse existido.
Gould aproveita esta idéia na forma de um experimento mental (sem a necessidade de um anjo) que consiste em “repassar o videotape (ou DVD ou Bluray, para os mais jovens) da vida novamente” e verificar como pequenas alterações afetariam o resto da evolução. Como as diversas possibilidades seriam afetadas caso certos eventos não tivessem ocorrido? Como grupos extintos poderiam alterar a atual configuração da vida caso algum (ou alguns deles) não tivessem sido extintos, por exemplo, por impactos de meteoros (ou grandes mudanças climáticas, vulcanismo, pandemias virais) ou, mesmo se, as mutações apropriadas tivesse ou não

ocorrido no tempo e na ordem correta?
Neste livro, Gould nos conta a história da estranhamente bela fauna fóssil, datada do período cambriano, achada no folhelho de Burgess (Burgess Shale) na Colúmbia Britânica, que havia sido 'redescoberta' por um time de paleontólogos e que revelava uma riquíssima variedade de planos corporais (para muitos) extremamente mais díspares (disparidade é, neste contexto, um termo técnico) do que os encontramos hoje. Este fato poderia significar que o número de possíveis filos (no caso de animais, filos são distinguidos pelos diferentes planos de organização corporal básica) no cambriano seria, na realidade, muito maior do que o atual, mesmo que a diversidade (em termos de número de espécies e linhagens) atualmente seja maior.
Filos inteiros, de aparência bastante alienígena para nós, teriam sido extintos, com isso reduzindo drasticamente a bio-disparidade existente. Assim, ao invés de uma árvore com uma copa larga e florida, a representação mais adequada da história da vida animal (em relação a disparidade morfológica) seria uma conífera, como um pinheirinho de natal. Hoje esta perspectiva em relação a diminuição da disparidade é posta em dúvida por vários cientistas. Assim como, também é questionada a idéia de que muitos destes filos extintos não teriam parentesco mais próximo com filos atuais. O que se acredita atualmente é que a maioria daqueles seres, na realidade, fossem formas basais, ainda não completamente características moderna do grupo, mas ainda assim aparentados com os filos 'descendentes' modernos (crown groups). Embora, as idéias mais radicais de Gould, como a idéia de que a disparidade da própria fauna de Burgess superava em muito a disparidade atual. Mas mesmo assim, hoje, após muitas avanços em técnicas de quantificação da 'disparidade', estes estudos revelaram que a disparidade, i.e. variedade de planos corporais, alcançou seu máximo já neste período inicial da evolução animal e só mais tarde foi seguida pela diversidade taxonômica, portanto, preservando algumas das intuições de Gould. O Paleobiólogo, Douglas Erwin, coloca a questão da seguinte maneira:
"Com as avaliações quantitativas da morfologia substituindo a contagem de taxa 'superiores' como uma métrica de disparidade morfológica, numerosos estudos demonstraram a construção rápida do morfo-espaço no início das radiações evolutivas, e enfatizou a diferença entre as medidas taxonômicas da diversidade morfológica e avaliações quantitativas de disparidade." (Erwin, 2007)
A questão principal, no entanto, é que um número enorme de animais bizarros foi extinta por algum processo contingente, e a vida animal atual é apenas uma fração bastante derivada deste momento da evolução dos seres multicelulares conhecida como 'explosão' cambriana. Imagine então, se estes fatores que causaram a extinção destas linhagens, em particular, não tivessem ocorrido. O quão diferente seria a vida atual? Será que uma cascata de eventos de especiação e extinção muito diferentes aconteceria? Será que ainda teríamos tido os dinossauros e mais tarde a explosão de diversificação de mamíferos, ambos grupos de vertebrados, membros do filo Chordata? Ou será que criaturas diferentes teriam surgido ocupando os mesmos habitats, porém de forma muito diferente e criando novos nichos? Ou ainda: Será que mesmo que criaturas diferentes evoluíssem (descendentes de linhagens extintas, diferentes das nossas linhagens parentais), estas teriam convergido em direção a formas semelhantes as das biotas modernas, talvez ocupando habitats muito parecidos, formando assim nichos equivalentes aos modernos? No momento, realmente não sabemos, apesar de suspeitarmos fortemente que (em um nível mais detalhado) muitas das criaturas e nichos atuais provavelmente não existiriam. Quem sabe, o nosso próprio nicho (generalistas de distribuição global com organização sociocultural extremante complexa, muito dependentes de tecnologia) não estaria entre os que, simplesmente, não teriam evoluído, sendo a evolução de criaturas inteligentes e capazes de nosso nível cultural e de tecnologia, um mero acidente, associado a vários eventos particulares e contingentes.
No entanto, observações recentes indicam que os genes não são todos iguais aos olhos da evolução. Mutações evolutivamente relevantes tendem a se acumular em genes conhecidos como hotspot e mesmo em posições específicas dentro destes genes. Assim a evolução genética é limitada pela função do gene, a estrutura das redes genéticas e pela biologia populacional.
A base genética da evolução pode ser previsível, pelo menos até certo ponto, e uma maior compreensão do presente exigiria a incorporação da previsibilidade das funções e características específicas de genes na teoria evolutiva (Stern e Orgogozo, 2009).
Desde o começo da moderna biologia evolutiva - quando Charles Darwin e Alfred Russell Wallace propuseram que a biodiversidade era basicamente resultado da seleção natural agindo sobre variações hereditárias nas populações – já havia o reconhecimento, tanto por Darwin quanto por Wallace, da importância da variação hereditária para a compreensão da evolução, mesmo que não fosse claro como a herança se daria. Apenas no início do século 20, com a 'redescoberta' dos estudos de Mendel, é que foi possível tratar matematicamente a dinâmica dos alelos nas populações, o que iniciou o campo da genética de populações.
Geneticistas de populações tratam genes e alelos (as variantes de um mesmo gene em um dado locus) como entidades genéricas, como 'partículas' herdadas e que de alguma forma causam variação na aparência, comportamento e fisiologia dos organismos, o que chamamos conjuntamente de fenótipo (Stern e Orgogozo, 2009).
Stern & Orgogozo chamam a atenção para um fato muito importante:
“Esse nível de abstração era adequado, já que uma compreensão molecular da função do gene estraria muitas décadas no futuro. Mesmo com essa visão rudimentar da função do gene, no entanto, a genética da populações esclareceu muito a forma como as populações evoluem, e essa compreensão teórica estimulou a 'nova síntese', a combinação genética de populações com a ecologia, sistemática e biogeografia para explicar e explorar as muitas questões em evolução.” (Stern & Orgogozo, 2009)
Acontece que nos últimos 40 anos, a biologia molecular têm elucidado, de forma cada vez mais detalhada, qual a participação dos genes na regulação de processos biológicos. Infelizmente, apenas algumas observações mecanicísticas mais básicas foram integradas à biologia evolutiva, como a distinção entre substituições 'sinônimas' (substituições de nucleotídeos que não alteram o aminoácido codificado) e 'não-sinônimas' (substituições de nucleotídeos que alteram o aminoácido codificado) em regiões gênicas codificadoras de proteínas. Porém, outros aspectos da biologia molecular atual contribuem muito pouco para a teoria evolutiva, mas como Stern e Orgogozo (2009) afirmam:
“O tempo chegou agora para integrar as especificidades da biologia molecular e desenvolvimento à biologia evolutiva. Nos últimos 15 anos, muitos exemplos de genes e mutações que causam mudanças evolutivas foram identificadas (1). Padrões nestes dados sugerem que uma síntese da biologia molecular do desenvolvimento com a teoria evolutiva irá revelar novos princípios gerais da evolução genética.” (Stern & Orgogozo, 2009)
Bem vindos a simpática 'Evo Devo'!
Em artigo da revista Science, Stern e Orgogozo (2009) discutem várias evidências que apontam para que a evolução genética seja, pelo menos parcialmente, previsível já que a arquitetura genômica (fruto da evolução pregressa) dos seres vivos coage e limita alguns caminhos evolutivos.
1) Distribuição não-aleatória das mutações evolutivamente relevantes:
“Estudos recentes sugerem que as mutações que contribuem para a variação fenotípica [mutações evolutivamente relevantes (2)] não são distribuídas aleatoriamente em todas as regiões genéticas. A evidência mais convincente vem de casos de evolução genética paralela: a evolução independente de alterações fenotípicas semelhantes em espécies diferentes devido às mudanças em genes homólogos ou às vezes na posição mesmo aminoácido de genes homólogos.” (Stern e Orgogozo, 2009)
Muitos casos de evolução paralela foram descobertos em todos os reinos. Seguem alguns exemplos:
1.a) Pelo menos 20 populações separadas da planta Arabidopsis thaliana têm evoluído mutações de codificação nulas (as mutações que eliminar completamente a função da proteína) no gene frigida que causa início do florescimento.
1.b) Resistência ao DDT e a piretróides evoluiu em 11 espécies de insetos a partir de mutações no gene que codifica, ou um ou outro, aminoácido, Leu1014 ou Thr929, que formam o canal de sódio dependentes da voltagem codificado pelo gene para.
1.c) Duas populações de vírus, submetidos, independentemente, à evolução experimental em um hospedeiro acumularam muitas das mesmas mutações de aminoácidos.
No total, são cerca de 350 mutações evolutivamente relevantes que têm sido encontradas em plantas e animais, e mais da metade desses casos representam casos de evolução genética paralela.
Stern & Orgogozo, então, afirmam:
“Uma explicação para a evolução genética paralela é que a maioria dos genes desempenha um papel especializado durante o desenvolvimento, e apenas alguns genes podem evoluir para gerar variantes fenotípicas particulares. Por exemplo, mutações na rodopsina podem alterar a sensibilidade à luz de diferentes comprimentos de onda (6), e as mutações no lisozima podem aumentar a atividade enzimática no pH especial de fermentação intestinal (7). Mas o inverso não seria verdadeiro. Mutações na rodopsina não são susceptíveis de melhorar a fermentação e as mutações em uma enzima digestiva não ajudam a detecção de um determinado comprimento de onda de luz, mesmo que cada proteína fosse expressa no órgão de reciprocidade.” (Stern e Orgogozo, 2009)
Em parte, a função do gene explica e evolução paralela, mas isso não é sempre verdade já que em vários casos os padrões observados de evolução genética paralela, podem ser alcançados por mutações em um grande número de genes semelhantes, capazes de produzir as mesmas alterações fenotípicas:
“Por exemplo, embora mais de 80 genes regulam o tempo de floração (8), alterações em apenas um subconjunto desses genes têm produzido mudanças evolutivas no tempo de floração (3). Centenas de genes regulam o padrão de muitas projeções epidérmicas, chamadas 'tricomas', em larvas de Drosophila melanogaster. Mas apenas um gene, chamado shavenbaby, evoluiu para alterar os padrões de tricomas das larvas entre as espécies Drosophila e este gene tem múltiplas mutações acumuladas evolutivamente relevantes (9).” (Stern e Orgogozo, 2009)
Então o que existe de especial sobre esses genes hotspots?
A resposta para esta questão encontram-se em grande parte na biologia do desenvolvimento. Inclusive, no caso particular de genes hotspots como shavenbaby, para 'o porquê' genes como eles existem. A estrutura dos genes eucarióticos e o controle espacial e temporal de sua expressão está na base deste processo. Abaixo pode se observar um esquema simplificado que ilustra a estrutura de um gene eucariótico.

Um conceito chave para compreendermos o desenvolvimento embriológico e, desta forma, a emergência e evolução dos planos corporais e das outras estruturas morfológicas, é o de 'vias de sinalização'. Para que um ser multicelular cresça e se desenvolva, como é mais freqüente, a partir de uma única célula, é que esta célula inicial deve se multiplicar de forma integrada e controlada, assim as diversas linhagens de células 'filhas' diferencie-se em células particulares e formem tecidos específicos, órgãos e estruturas através de migração, movimentos coletivos e deformações conjuntas que geram a forma final do ser multicelular que está se desenvolvendo.
Estes processos dependem da habilidade das células 'comunicarem-se' entre si e com seu meio adjacente, alterando seu comportamento de acordo com estas informações contextuais podendo assim contribuir para a formação de um todo auto-organizado. Esta comunicação se dá através da troca de sinais químicos entre as células (por exemplo, através de moléculas difusíveis de curta e longa distância, como secreções parácrinas e hormônios) ou do contato direto entre células através do entre moléculas de superfície, proteínas transmembrana cujos domínios extra-celulares funcionam como sistemas receptores-ligantes, como ocorre com as
chamadas moléculas de adesão celular. Estas mensagens químicas, e outras 'pistas' de contexto, ao interagir (por exemplo, ao ligarem-se com um proteína receptora de membrana) com as células-alvo, produzem mudanças nos estados bioquímicos internos destas células, inclusive nos seus estados de ativação genômica. Isto é, genes são silenciados e expressos ('ligados' e 'desligados') ou tem seus níveis de expressão aumentados ou diminuídos como conseqüência destes sinais. Estas mudanças, por sua vez, podem induzir estas células a multiplicar, migrar, diferenciar-se, aumentar de tamanho, mudar de forma, morrer (a chamada morte celular programada ou apoptose), liberar outras moléculas de sinalização ou expressar proteínas diferentes em suas membranas. Os eventos bioquímicos intracelulares que servem de intermediários para esta comunicação constituem-se nas 'vias de sinalização' e nos mecanismos de 'transdução de sinal'. São por estas vias bioquímicas que muitos biólogos evolutivos tem se interessado nas últimas décadas, como forma de explicar a evolução morfológica através de modificações nos sistemas de controle do desenvolvimento.
Os processos de transdução de sinal começam com a ligação de uma molécula, um ligante (difusível ou um
domínio extracelular de uma proteína de membrana de uma célula vizinha), a um receptor de membrana. Isto é, uma proteína transmembrana que é estimulada a mudar de conformação ao interagir com o seu ligante, no caso a molécula sinalizadora, induzindo uma cascata de eventos bioquímicos como a produção de segundos
mensageiros (como DAG e IP3), hidrolisação de ATP, ativação de proteínas fosforiladoras chamadas de proteínas quinases, abertura de canais iônicos, liberação de Ca++, indução ou inibição da transcrição de genes e conseqüente síntese (ou bloqueio) de novas proteínas.
Um dos elementos cruciais deste processo são os chamados elementos Cis-regulatórios e os fatores Trans-ativos, unidades essenciais no controle da transcrição gênica que tem ocupado muitas das pesquisas em genética do desenvolvimento e genética evolutiva. Os primeiros (elementos Cis-) são seqüencias de DNA que modulam a transcrição do genes a que estão associadas. Já os fatores Trans- são os elementos protéicos que ligam-se as estas seqüencias Cis- modulando-as.
Durante o desenvolvimento, várias vias de sinalização celular e fatores de transcrição agem conjuntamente para dividir progressivamente o embrião em um mapa virtual que especifica quando e onde irão se formar os órgãos (Stern e Orgogozo, 2009). As interações entre os genes que codificam estas moléculas de sinalização e os fatores de transcrição podem ser representadas como uma rede genética. Interações entre genes são moduladas em grande parte pelas regiões cis-regulatórias (veja esquema a direita e abaixo - Clique a figura para aumentá-la).
A estas regiões Cis-regulatórias ligam-se fatores de transcrição, e os efeitos somados de destes fatores em um gene-alvo determinam se o gene é expresso ou não. Genes controladores de padrões agem dentro de complexas redes genéticas e, normalmente, cada gene contribui para a formação de padrões de desenvolvimento em vários tipos de células. Por exemplo, os genes controladores de padrões que são mais ativos durante o desenvolvimento embrionário da epiderme contribuem também para o desenvolvimento dos sítios de inserção de músculos, órgãos sensoriais, poço traqueal, tricomas, ou outros tipos celulares. Nesta rede regulatória de desenvolvimento genes controladores de padrões primeiro colaboram para dividir a epiderme embrionária em domínios que expressam fatores de transcrição distintos. Estes genes controladores de padrões, então, regulam a expressão do gene shavenbaby, que funciona como um sistema de controle de 'input-output' ('entrada-saída'). Estes genes de 'entrada-saída', funcionam como nós de uma rede causal, integrando as informações espaço-temporais complexas (a entrada) e ativando todo um 'programa' de diferenciação celular (a saída) (Stern & Orgogozo, 2009).
A proteína Shavenbaby ativa a expressão de uma bateria de genes-alvo que transformam uma célula epidérmica em uma célula de tricomas. Cada gene alvo desencadeia um aspecto específico de diferenciação celular e a produção de tricomas diferenciados requer uma expressão coordenada de todos os genes-alvo. O padrão de tricomas por todo o corpo é, assim, determinada pela distribuição da proteína Shavenbaby na epiderme, que é controlada pela região cis-reguladora do gene shavenbaby (Stern & Orgogozo, 2009):
“O gene shavenbaby serve como um elo para as informações de formação de padrões fluírem para dentro e para a informação dos destinos celulares fluírem para fora.” (Stern & Orgogozo, 2009)

O ponto que Stern & Orgogozo querem enfatizar é que, em toda a rede de regulação gênica do desenvolvimento dos embriões de drosófila, apenas um gene, no caso shavenbaby, por sua posição nodal na
rede de sinalização e função muito especializada de integrar 'o módulo da morfogênese tricomas', pode acumular mutações que alteram os padrões de tricomas sem perturbar outros processos de desenvolvimento. Assim, alterações em genes desta mesma via de desenvolvimento, porém a cima na hierarquia de sinalização, irá alterar a produção de tricomas, mas estas mutações também perturbarão outros órgãos. De forma complementar, alterações em qualquer um dos genes a baixo, na hierarquia da rede, não serão suficientes para criar ou eliminar um tricoma; alterações em concerto em vários genes 'abaixo' são necessárias para construir um tricoma (Clique nas figuras para aumentá-las).
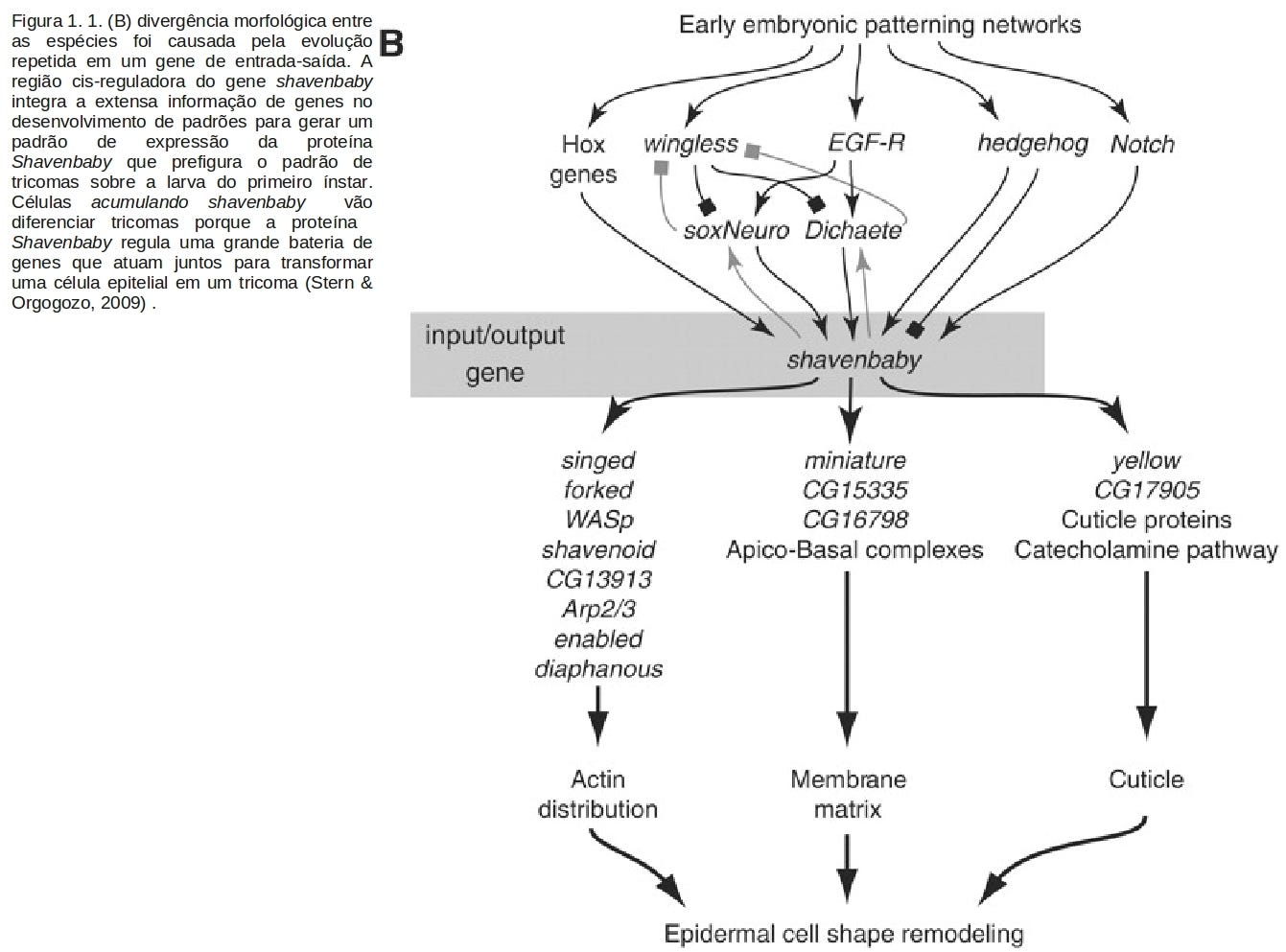
Além disso, Stern & Orgogozo nos lembram, que todas as mutações evolutivamente relevantes em shavenbaby, que foram identificadas até o momento, alteram a região cis-regulatória e não a região codificadora da proteína. Mutações na região codificadora da proteína que alteram a função shavenbaby em todas as células em que a proteína shavenbaby se acumula, iriam alterar todos os tricomas produzidos em larvas e adultos (Stern & Orgogozo, 2009).
“Assim, em uma perspectiva desenvolvimental, esclarece por que shavenbaby é um hotspot para mutações evolutivamente relevantes e por que essas mutações ocorrem na região cis-reguladora do gene. Nós prevemos
que as regiões cis-regulatórias de outros genes de 'entrada-saída' podem ser hotspots para outras características fenotípicas.”
“O gene shavenbaby fornece um exemplo de um princípio mais geral: que as mutações que afetam várias características fenotípicas, as chamadas mutações pleiotrópicas, não são susceptíveis de contribuir para a evolução adaptativa. Como discutiremos a seguir, pleiotropia e outros parâmetros genéticos e da genética de
populações parecem influenciar a distribuição das mutações evolutivamente relevantes.” (Stern & Orgogozo, 2009)
2. Os fatores que influenciam a distribuição de mutações evolutivamente relevantes:
2.1 Pleiotropia:
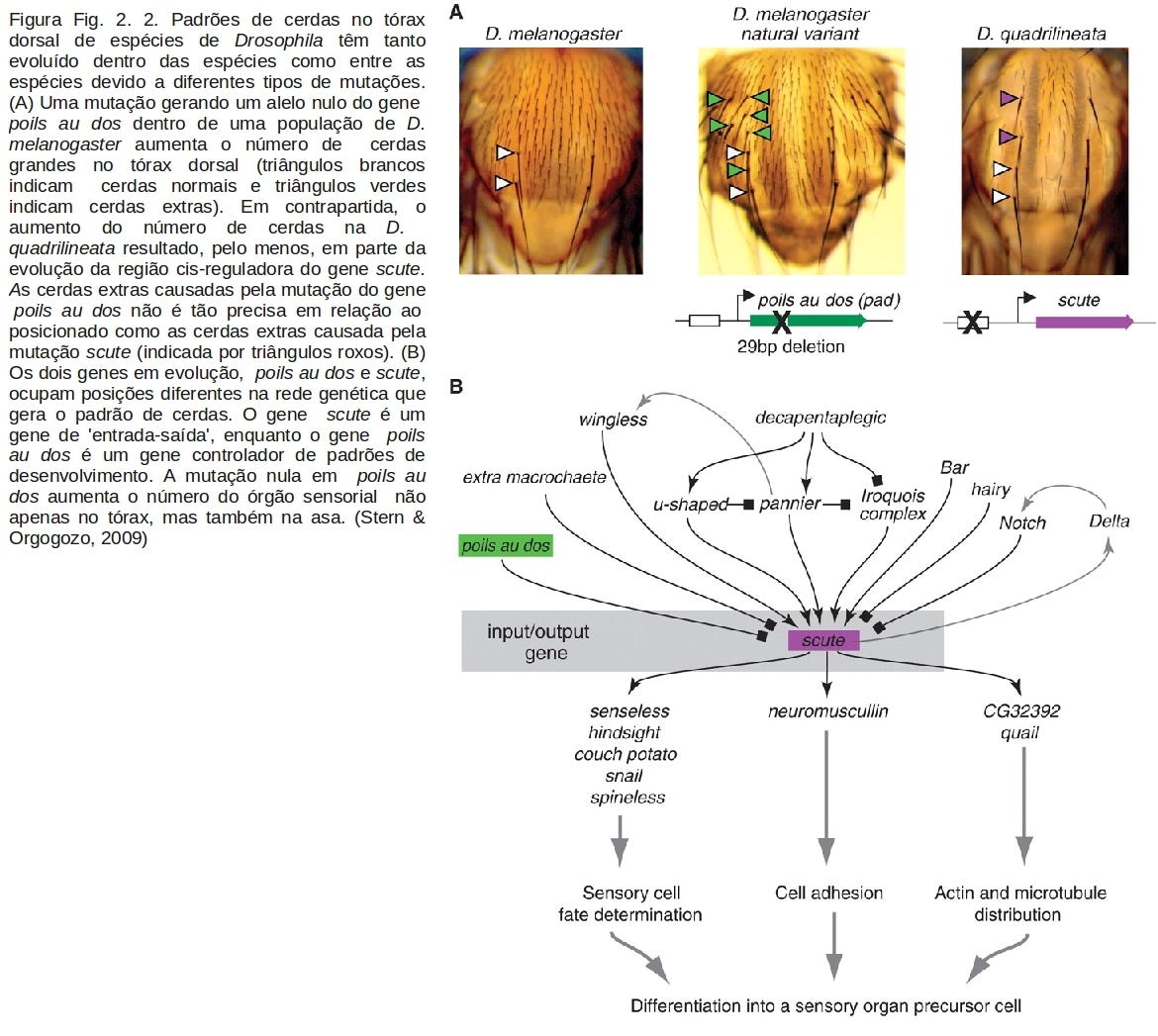
Mutações pleiotrópicas alteram várias características aparentemente não relacionados ao mesmo tempo. Duas mutações que causam aumento evolutivo do número de cerdas torácicas na drosófila ilustram a diferença entre mutações com efeitos específicos e pleiotrópicos (Fig. 2):
“Uma mudança em uma seqüencia cis-reguladora do gene scute afeta o número de órgãos sensoriais apenas sobre o tórax (12), enquanto que uma mutação na codificação dos gene poils au dos aumenta o número de órgãos sensoriais no tórax e nas asas (13). Mutações sobre poils au dos são mais pleiotrópicas que mutações sobre as regiões cis-regulatórias do gene scute. Scute, shavenbaby são genes do tipo 'entrada-saída', enquanto poils au dos é um gene de controle de padrões que, em conjunto com outro genes, regula a expressão scute (Fig. 2). Mutações, com efeitos pleiotrópicos, raramente mudam todas as características fenotípicas de uma forma favorável, e evidências experimentais indicam que os efeitos pleiotrópicos tendem a reduzir a aptidão (14). Seleção pode favorecer cerdas extras no tórax, mas não em órgãos como a asa. Mesmo se um efeito de uma mutação pleiotrópica induzisse uma grande melhoria na condição física, outros efeitos podem ser deletérios e reduzir a probabilidade de que a mutação se estabeleça na população (15).” (Stern & Orgogozo, 2009)
2.2 Epistasia:
A epistasia é um fenômeno também particularmente importante e é evidenciado por uma observação bastante simples. Quando examinamos os efeitos de uma mutação sobre um único fundo genético (os demais genes, e variantes alélicas, que compõem o genoma afetado em questão), esta mutação pode ter um efeito específico ou mesmo pleiotrópico, mas, em outro fundo genético, a mesma mutação pode produzir um efeito fenotípico diferente por causa das interações alélicas não aditivas, e é isso que chamamos 'epistasia'. São os efeitos da interação entres os produtos de genes diferentes que não podem ser reduzidos a um soma simples de seus efeitos individuais (Stern & Orgogozo, 2009).
“Por exemplo, um alelo de A. thaliana aumenta o crescimento em um fundo genético, mas reduz o crescimento em um fundo genético diferente (16). O segundo fundo genético não é simplesmente deletério, em geral, pois um alelo variante em um segundo locus provoca maior crescimento neste fundo. Assim, os efeitos de uma mutação podem depender da variação genética presente em outros loci.” (Stern & Orgogozo, 2009)
Epistasia é extremamente comum em populações naturais e, por vezes, podem reduzir a taxa de evolução, aumentando a variância associada a uma mutação em particular, causando efeitos flutuantes de aptidão nas
mutações tornando estes efeitos dependentes da herança genética de cada indivíduo:
“Assim, em uma população de Arabidopsis contendo múltiplas origens genéticas, esperamos que a seleção para tamanho maior tenderá a favorecer alelos não-epistáticos que aumentem o crescimento em todas as fundos genéticos, em vez de alelos epistáticos que aumentem o crescimento em apenas um fundo genético.” (Stern & Orgogozo, 2009)
2.3 Plasticidade:
Populações expostas a repetidas alterações ambientais podem evoluir mecanismos genéticos que produzem diferentes fenótipos adaptados a diferentes condições ambientais: chamada de plasticidade fenotípica.
“Por exemplo, os pulgões podem produzir múltiplas formas fenotípicas em resposta às condições ambientais, incluindo as formas assexuadas que se reproduzem rapidamente e formas sexuadas que depositam ovos hibernantes. As mutações que eliminam as formas sexuadas que reduzem a plasticidade podem dar, a uma linhagem, uma vantagem a curto prazo, uma taxa muito mais rápida de reprodução. Mas, a longo prazo, as linhagens de pulgões que não produzem formas sexuadas tendem a se extinguir, talvez porque eles não consigam se adaptar às novas condições ambientais.” (Stern & Orgogozo, 2009)
Algo semelhante ocorre em A. thaliana com o gene frigida que controla a plasticidade para o tempo de floração. Frigida responde a baixas temperaturas para induzir a floração. Em regiões com invernos quentes, mutações nulas em frigida podem fornecer um benefício de curto prazo de forma consistente, ao desencadear o florescimento, mesmo na ausência de um inverno frio. Mas estas mutações eliminam a plasticidade para época de floração, possivelmente prevenindo estas plantas de se adaptar a temperaturas mais frias ou de recolonizar áreas em climas mais frios. Assim, a abundância de mutações nulas em frigida em populações de Arabidopsis deve resultar de fatores que compensam as conseqüências negativas da plasticidade reduzida (Stern & Orgogozo, 2009).
2.4 Força de seleção:
Quando uma mudança de ambiente favorece o fenótipo que é muito diferente do fenótipo médio em uma população, mutações que causam grandes alterações fenotípicas para o novo 'ideal' serão favorecidas, pelos menos inicialmente (Stern & Orgogozo, 2009).
Por exemplo, raças domesticadas recentemente experimentaram provavelmente forte seleção por parte dos criadores, e muitos traços, típicos de domesticação recente, são resultantes de mutações que causam grandes efeitos fenotípicos, incluindo efeitos pleiotrópicos deletérios. Como exemplo, seis diferentes tipos de mutações nulas que interferem diretamente com a codificação do gene da miostatina causam hipertrofia muscular em diferentes raças de bovinos .
A Miostatina é um membro da superfamília de fatores de crescimento transformadores-β, atuando como regulador negativo do desenvolvimento muscular. Embora mutações nulas no gene da miostatina gerem animais com mais carne e menos gordura, estes animais têm dificuldades no parto e reduzida tolerância ao estresse. Isso indica que forte seleção artificial, durante a domesticação podem, obviamente, superar os efeitos negativos pleiotrópicos das mutações nulas no gene da miostatina (Stern & Orgogozo, 2009).
2.5 História da População:
O artigo de Stern & Orgogozo enfatiza um outro determinante importante da evolução genética, o tamanho das populações, passadas e atuais (Para maiores detalhes veja aqui). Estes influenciam em muito a evolução genética, sobretudo a eficiência da seleção natural, já que tamanhos pequenos de populações aumentam os efeitos da amostragem aleatória dos alelos, aquilo que os geneticistas chamam de deriva genética aleatória e já foi discutido, em outras ocasiões, aqui no evolucionismo:
“Em pequenas populações, a deriva genética permitirá que alelos deletérios ocasionalmente aumentem em freqüência. Por exemplo, uma pequena população pura de beduínos em Israel desenvolveu uma alta freqüência de um alelo recessivo que provoca surdez (20). Com a forte deriva genética em pequenas populações, a seleção natural vai deixar de promover a disseminação de mutações adaptativas de pequeno efeito. Em vez disso, em comparação com grandes populações, mutações adaptativas de efeito relativamente grandes tendem a evoluir através da seleção natural em populações pequenas.” (Stern & Orgogozo, 2009)
Pequenas populações também têm um outro efeito crítico sobre a evolução. Elas limitam o número total de novas mutações introduzidas em uma população a cada geração. Logo, populações pequenas podem acabar ficando relegadas a mutações, longe das 'ideais' (com conseqüências pleiotrópicos e epistáticos) simplesmente porque as mutações potencialmente superiores ocorrem a uma taxa inferior e são perdidas pelos efeitos da deriva genética (Stern & Orgogozo, 2009).
A abundância de mutações nulas no gene frigida em populações de A. thaliana destaca a importância da história na evolução genética das populações:
“Mutações nulas em frigida tem a conseqüência negativa de reduzir a plasticidade para o tempo de floração. Essas mutações também têm efeitos pleiotrópicos [reduzem a produção de frutos (21)] e mostrar epistasia em relação a outros genes que controlam o tempo de floração (22).” (Stern & Orgogozo, 2009)
Stern & Orgogozo (2008, 2009), afirmam, então que 'estas observações sugerem que as mutações nulas em frigida não refletem os 'alelos ideais' para controle o tempo de floração'. Para os pesquisadores, mutações nulas neste gene (frigida) apenas raramente, se é que alguma vez, devem estar envolvidas na divergência fenotípica entre as espécies, mas a seleção natural superou os efeitos deletérios das mutações nulas em frigida e promoveu a difusão dessas mutações em populações pequenas. As subpopulações de A. thaliana que migraram da Escandinávia, aparentemente, seguindo as pegadas da agricultura ao redor do mundo, se adaptaram às condições locais, incluindo os invernos relativamente quentes e mais curto de regiões temperadas, o que foi facilitado pelo fato destas plantas serem auto-fertilizadas, por isso mesmo uma única planta pode dar origem a uma nova população. Assim, estas subpopulações pequenas forneceriam poucas oportunidades para mutações benéficas com efeitos específicos, e não-pleiotrópicos, aparecerem, assim a seleção acabou por favorecer mutações de maior efeito, tais como mutações nulas como as que ocorrem em frigida. Desta forma Stern & Orgogozo (2009) concluem que 'a abundância de mutações nulas em frigida provavelmente reflete o fato de que estas mutações ocorrem a uma taxa superior à mutações associadas sem conseqüências deletérias'.
A base genética da evolução de curto e longo prazo:
O exemplo frigida não é o único. Em muitas plantas e animais, a evolução ao longo de períodos extensos de tempo (variação entre espécies) parece diferir de várias formas da evolução em períodos mais curtos de tempo(variação entre raças domesticadas e entre indivíduos dentro de uma espécie) . Aqui estão três maneiras
gerais em que a evolução genética a longo prazo e de curto prazo são diferentes (Stern & Orgogozo, 2009).
Primeiro, a epistasia é comumente encontrada em mutações que contribuem para a variação fenotípica dentro das espécies, que é raramente observada para as mutações que causam as diferenças entre as espécies. Dentro de D. melanogaster, a variação no número de cerdas é causada por múltiplos loci de efeito relativamente pequeno, e esses loci têm efeitos epistáticos da mesma ordem de grandeza que os efeitos aditivos. Em contrapartida, as diferenças morfológicas entre as espécies Drosophila são devidas a múltiplos loci de efeito pequeno para intermediário que raramente mostram epistasia. Estudos de variação do tamanho corporal em frangos mostram um padrão semelhante, com alelos segregantes dentro das espécies mostrando mais epistasia que os alelos envolvidos na diferenciação das espécies (Stern & Orgogozo, 2009).
Por outro lado, mutações nulas que surgem com freqüência, e muitas vezes causam efeitos pleiotrópicos e epistáticos, parecem contribuir mais para a variação fenotípica dentro das espécies do que as diferenças fenotípicas entre as espécies. Cerca de 55% das 99 mutações conhecidas por causar traços que tornam animais e plantas mas adequados a domesticação são mutações nulas que atingem as regiões de codificação dos genes, enquanto apenas 7% dos 75 mutações conhecidas para causar diferenças interespecíficas são mutações nulas envolvendo a codificação (Fig. 3). Por exemplo, embora os rebanhos domésticos de gado evoluíram múltiplas vezes mutações nulas do gene da miostatina, todas as espécies de mamíferos estudados até o momento possuem um gene da miostatina funcional (Stern & Orgogozo, 2009).
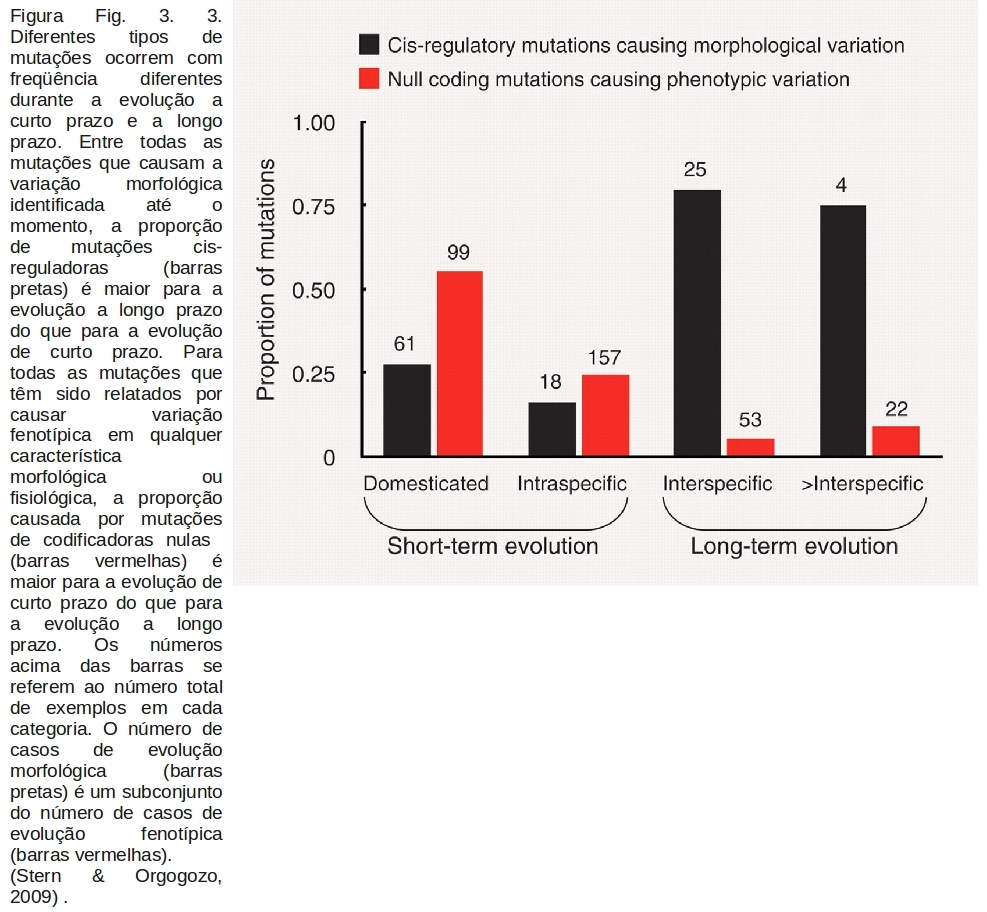
Em terceiro lugar, a proporção de mutações cis-regulatórias causando variação morfológica difere entre os níveis taxonômicos. As mudanças em características morfológicas podem se dar através de alterações em regiões codificadoras ou através de alterações nas seqüencias cis-regulatórias (Fig. 2). Como as mutações em regiões cis-regulatórias, muitas vezes, têm menos efeitos pleiotrópicos do que mutações em regiões codificantes, é esperado que as alterações morfológicas envolvam principalmente mutações nessas seqüencias. Dentro das espécies, a maioria das mutações que causam a variação morfológica encontradas ocorriam em regiões codificadoras de proteínas (Fig. 3). Em contraste, entre as espécies, mais mutações que causam diferenças morfológicas foram encontradas em regiões cis-regulatórias. Presumivelmente, muitas das mutações em regiões codificadoras encontradas dentro de uma espécie não se espalham através das populações, talvez por causa dos efeitos pleiotrópicos deletérios (Stern & Orgogozo, 2009). Então as pressões seletivas podem evitar ou dificultar a fixação deste tipo de mutação.
Estas diferenças notáveis e inesperadas entre a evolução genética, de curto e longo prazo, só recentemente acumularam-se em um número suficiente de estudos de casos para adquirirem relevância científica. Esses padrões são coerentes com as expectativas teóricas de como os cinco parâmetros discutidos anteriormente (pleiotropia, epistasia, plasticidade, resistência da seleção, e estrutura da população) devem influenciar a evolução da genética.
Evolução durante longos períodos, refletida nas diferenças entre as espécies, deve resultar de mutações relativamente livres de efeitos pleiotrópicos e epistáticos.
Em contraste a evolução, em períodos mais curtos, refletida nas diferenças entre as raças domesticadas e na variação dentro da espécie, pode muitas vezes resultar de mutações que perturbam plasticidade ou que tenham efeitos pleiotrópicos e epistáticos.
Em resumo, as diferenças entre as espécies são causadas por um subconjunto parcial das mutações que surge no seio das populações naturais (Stern & Orgogozo, 2009).
Conclusões:
Embora acreditemos, normalmente, que as mutações ocorram de forma aleatória (para maiores detalhes sobre a questão da aleatoriedade nas mutações veja aqui) no genoma, a distribuição das mutações que causam a diversidade biológica parece ser altamente não randômica. Os efeitos evolutivamente relevantes de uma mutação, que contribuem para a evolução fenotípica, dependem, portanto, de todo um conjunto de questões genéticas, mais especialmente:
Função do gene;
Estrutura do gene;
Papéis dos genes e de seus produtos
Assim, para algumas mudanças fenotípicas (associadas às mutações evolutivamente relevantes), espera-se que estas se acumulem em alguns genes hotspot e até mesmo em determinadas regiões dentro destes genes individuais. Além disso, a biologia populacional e ecologia influenciam o espectro de mutações evolutivamente relevantes. Durante curtos períodos, as mutações adaptativas com efeitos pleiotrópicos deletérios poderão ser selecionadas por que as mutações 'próximas ao ideal' (i.e. sem efeitos deletérios) ainda não apareceram. Em contraste, durante longos períodos, mutações adaptativas sem efeitos pleiotrópicos deletérios têm mais oportunidades para surgir e serem assim selecionadas (Stern & Orgogozo, 2009).
Estas constatações levaram Stern & Orgogozo (2009) a sugerir que a base genética da evolução fenotípica, portanto, parece ser de certa forma previsível, mas estes fazem algumas ressalvas importantes que merecem atenção:
“Esses padrões que emergem na distribuição de mutações que causam modificações na diversidade fenotípica, são derivados a partir de um conjunto limitado de dados que foram extraídos da literatura, portanto, é possível que esses padrões reflitam tendenciosidades no modo como os cientistas têm procurado por mutações evolutivamente relevantes (1). Por exemplo, muitos pesquisadores focam em genes candidatos, o que impede a descoberta de genes previamente desconhecidos. No futuro, esperamos que a adoção generalizada de abordagens experimentais imparciais, como o mapeamento genético, por exemplo, irão fornecer dados para testes robustos da previsibilidade da evolução genética.” (Stern &Orgogozo, 2009)
O mapeamento genético pode ser realizado dentro de cada espécie e, em casos raros, entre espécies estreitamente relacionadas através de cruzamentos controlados e análise de QTL (Quantitative Trace Locus) das características de híbridos com diferentes fundos genéticos. Como alternativa, a substituição de todos os genes de uma espécie elos de uma segunda espécie, gene por gene, embora experimentalmente entediante, pode permitir inquéritos imparciais para as espécies que não podem ser cruzadas. Esta abordagem permitiria comparações entre táxons distantemente relacionados e forneceria um teste direto para revelar se os táxons distantemente relacionados acumulam diferentes tipos de mutações evolutivamente relevantes que as espécies estreitamente relacionadas (Stern & Orgogozo, 2009).
Espera-se que mais previsões quantitativas precisas, sobre as mutações responsáveis pela evolução fenotípica, venham desta síntese adicional da biologia molecular do desenvolvimento e da genética populacional. Novos modelos teóricos provavelmente abordarão vários parâmetros de genética de populações, em um quadro de referência que leve em conta a estrutura genômica e organização desenvolvimental no qual estes parâmetros estão inseridos. Estes modelos podem ajudar a explicar como a distribuição das mutações, surgidas espontaneamente, se traduz na distribuição de mutações e efeitos mutacionais em populações segregantes e como estes processo influenciam a evolução em curto e longo prazo (Stern & Orgogozo, 2009).
O fato de que a evolução genética possa, a longo prazo, representar apenas um subconjunto parcial das mutações tem várias conseqüências práticas. Entre elas podemos citar o desenvolvimento de algoritmos computacionais mais eficientes - que utilizem as estratégias de busca evolutiva, para a melhoria das colheitas agrícolas e criação de animais, já que a domesticação frequentemente seleciona mutações efeitos pleiotrópicos deletérios – juntamente com a ajuda aos engenheiros e criadores na exploração das classes de mutações que parecem estar em jogo na evolução de longo prazo (que envolve a seleção de mutações efeitos fenotípicos específicos e que minimizem efeitos pleiotrópicos e epistáticos), o que pode facilitar a escolha de raças que possuam características desejáveis sem propriedades associadas desfavoráveis.
No entanto, de um ponto de vista teórico mais abrangente, estes fatos nos permitem uma melhor apreciação da importância dos processos de desenvolvimento na evolução biológica. As seqüencias Cis- e fatores Trans- são apenas a ponta do iceberg dos sistemas epigenéticos de controle do desenvolvimento. Mas deixemos estas questões para um próximo artigo.
____________________________________________________________________
Referências:
Stern, D., & Orgogozo, V. (2009). Is Genetic Evolution Predictable? Science, 323 (5915), 746-751 DOI: 10.1126/science.1158997
Stern, D., & Orgogozo, V. (2008). THE LOCI OF EVOLUTION: HOW PREDICTABLE IS GENETIC EVOLUTION? Evolution, 62 (9), 2155-2177 DOI: 10.1111/j.1558-5646.2008.00450.x
Referências adicionais:
Cleland, Carol E. (2001). “Historical science, experimental science, and the scientific method,” Geology 29, pp. 987-990.
Cleland, Carol E. (2002). “Methodological and Epistemic Differences Between Historical Science and Experimental Science,” Philosophy of Science 69, pp. 474-496.
Gould, Stephen Jay (1990) VIDA MARAVILHOSA - O acaso na evolução e a natureza da história (Tradução: Paulo César de Oliveira) Companhia das Letras ISBN 9788571641419 - http://www.companhiadasletras.com.br/detalhe.php?codigo=10228
Erwin, D.H. 2007. Disparity: Morphological patterns and developmental context, Palaeontology, 50:57-73.
Haack, Susan Inquiry and advocacy, fallibilism and finality: culture and inference in science and the law Law, Probability and Risk (2003) 2, 205–214
Lipton, Peter (2000)Inference to the Best Explanation, in W.H. Newton-Smith (ed.), A Companion to the Philosophy of Science, Blackwell 184-193.
Mosher DS, Quignon P, Bustamante CD, Sutter NB, Mellersh CS, Parker HG, Ostrander EA. A mutation in the myostatin gene increases muscle mass and enhances racing performance in heterozygote dogs. PLoS Genet. 2007 May 25;3(5):e79. Epub 2007 Apr 30. PubMed PMID: 17530926; PubMed Central PMCID: PMC1877876
Créditos figuras:
RICHARD BIZLEY / SCIENCE PHOTO LIBRARY
CHRISTIAN JEGOU PUBLIPHOTO DIFFUSION/ SCIENCE PHOTO LIBRARY
ALAN SIRULNIKOFF / SCIENCE PHOTO LIBRARY
LIBRARY OF CONGRESS / SCIENCE PHOTO LIBRARY
LAWRENCE BERKELEY NATIONAL LABORATORY/ SCIENCE PHOTO LIBRARY
EYE OF SCIENCE / SCIENCE PHOTO LIBRARY
DR JEREMY BURGESS / SCIENCE PHOTO LIBRARY
Based on PDB 1CUL http://commons.wikimedia.org/wiki/File:GPCR_mechanism.png (Original uploader was Bensaccount at en.wikipedia).
PDB 1rm1 EBI.jpg http://commons.wikimedia.org/wiki/File:PDB_1rm1_EBI.jpg (auhor: Jawahar Swaminathan and MSD staff at the European Bioinformatics Institute).
