Genomas e mais genomas em abril de 2013: Parasitas e Tartarugas
O mês de abril foi um mês agitado para a divulgação dos resultados de sequenciamentos de genomas animais. Além da publicação, no dia 18 de abril, dos resultados do rascunho do genoma da Latimeria chalumnae, o moderno 'Celacanto', abordado em post anterior, outros dois artigos relatando análises de genomas de outros animais, respectivamente, nas revistas Nature e Nature Genetics, também foram publicados.
O primeiro deles, publicado no dia 4 de abril na Nature, apresenta os resultados de comparações das sequencias genômicas de quatro vermes da ordem Cestoda, do grupo dos platelmintos, todos de grande interesse biológico e médico [1].
Já o outro artigo, publicado no dia 28 de abril, nos brindou com comparações dos rascunhos dos genomas de duas espécies de tartarugas e com uma análise comparativa dos padrões de expressão gênica de uma delas, a tartaura de casco mole, comprando-os com o desenvolvimento embrionário de pintos, fornecendo-nos pistas incríveis da história evolutiva deste grupo e sobre quais modificações resultaram na origem de seus característicos cascos [ 2].
As 'tênias', são platelmintos pertencentes a classe Cestoda, sendo conhecidos por causarem várias doenças. Este grupo de vermes parasitas é um dos mais antigos que temos notícia, sendo responsável por duas, das dezessete, doenças tropicais mais negligenciadas no mundo, de acordo com a OMS [1]. Embora os vermes adultos, que vivem nos intestinos de seus hospedeiros, sejam relativamente bem controláveis e até elimináveis, a infecção pelas larvas destes vermes resulta na formação de cistos nos tecidos e órgãos do hospedeiro, incluindo o sistema nervoso central, que podem ter consequências graves e são de dificíl tratamento. As larvas das tênias podem persistir sem causar sintomas em um hospedeiro humano por décadas a fio [1], eventualmente, causando várias complicações como a cegueira, epilepsia e mesmo a morte. Infelizmente, muitas vezes, quando os problemas são identificados, as complicações já encontram-se em estágios avançados e a cirurgia para a remoção dos cistos não é mais uma opção terapêutica viável [1].
Entre as adaptações morfológicas ao estilo de vida parasitários destes animais estão a ausência de intestino, da cabeça e de órgãos sensíveis à luz, além de possuírem uma superfície corporal resistente aos ácidos estomacais e a bílis dos hospedeiros, mas que é, ainda assim, capaz de absorver nutrientes [1]. O esforço de sequenciamento foi empreendido por uma colaboração internacional de pesquisadores de diversos centros de pesquisa, liderada pelo instituto Sanger. Eles sequenciaram o genoma completo, contendo cerca de 11000 genes, de quatro espécies deste grupo - os parasitas humanos, Echinococcus multilocularis, E. granulosus e Taenia solium e a espécie modelo, Hymenolepis microstoma - analisando também seus padrões de expressão gênicos [Veja matéria do site do Natural History Museum]. Foram encontardas várias evidências de adaptações específicas ao parasitismo [1]
Os genomas destes, variando de 115 a 141 megabases, mostram que os padrões de sintenia, ou seja, a ordem e posição relativa de genes e outras regiões genômicas, foi mantida, mesmo em relação a outros vermes, mais distantemente relacionados, como trematados, do gênero Schistosoma. Porém, vários genes ubíquos em outros animais, foram perdidos nos vermes cestodos, incluindo 34 famílias de genes que codificam proteínas homeobox (família de fatores de transcrição associados com a padroninzação dos planos corporais dos animais), além dos genes de várias vias de sinalização que determinam os destinos das células-tronco. Entre os outros fatos revelados pelo estudo estão, a posse por parte das tênias, de vias bioquímicas especializadas na desintoxicação e metabolismo bem ajustadas para os nutrientes obtidos dos seus hospedeiros, além de expansões específicas para cada espécie dos genes que codificam certas proteínas de choque térmico e de famílias de genes que codificam antígenos conhecidos. Estas descobertas têm também um impacto prático, já que o conhecimento do genoma destes animais possibilitou a identificação de novos potenciais alvos terapêuticos, incluindo alguns que podem ser afetados por produtos farmacêuticos já existentes [1], como podemos ver na tabela do artigo.
Abaixo e a direita, [figura retirada de Tsai et al., 2013] podemos observar uma filogenia dos principais ramos dos clado Bilateria, com os ecdysozoa (que incluem as moscas de fruta e os nematoides), os deuterostômios (incluindo o Anfioxo, peixes-zebra, camundongos e seres humanos), e lophotrochozoa (que incluem os platelmintos). Entre os ganhos e perdas de características ligadas aos ciclo de vida destes parasitas platelmintos, estão a evolução do endoparasitismo (a), a transmissão passiva entre hospedeiros (b), a aquisição de hospedeiro intermediário vertebrado (c), a capacidade de proliferação assexuada no hospedeiro intermediário (d). Já entre as características morfológicas que evoluíram estão a perda das 'taças óticas' (e), o ganho do epitélio sincicial típico dos neodermatos (f), a perda dos intestinos (g), a segmentação do corpo (h), além de alterações na camada de laminar (para conter um tipo de glicoproteínas, as apomucinas, especializadas: i). Em relação aos ganhos e perdas de características genômicas podem ser citadas o splice-líder trans-splicing* (1), a perda dos genes da via Wnt (2), a perda de NEK quinases, da biossíntese de ácidos graxos e dos genes ParaHox (3), da capacidade metabólica anaeróbia através da via da dismutação do malato/rodoquinona, fusão dos genes das enzimas glutarredoxina redutase e tiorredoxina redutase, formando a thioredoxin glutationa redutase (TGR) (4), a evolução da família Argonauta (Ago), Tênia e Schistossoma específica, de genes de microexons (MEGs) e da PROF1 GPCRs (um tipo de porteina receptora acoplada a proteína G) (5), a perda de genes dos peroxissomos (6), e a perda completa dos genes vasa, tudor e PIWI, dos genes da via do fator de transcrição NF-kB, a perda de 24 famílias de genes homeobox (indicado pela letra "H"), e das proteases envolvidas no metabolismo e na biossíntese de aminoácidos (7). Em tênias, os ganhos e perdas de características genômicas incluem inovação da distribuição bimodal de introns e novos transportadores de ácidos graxos (8), a expansão do gene da enzima glutationa S-transferases da classe Mu, dos antígenos GP50 e das tetraspaninas (9), a perda da via de biossíntese da molibdopterina, perda de 10 famílias de genes homeobox (10), menor quantidade de GPCRs e menos neuropeptídeos codificados por cada protopeptidio (11), além da expansão de proteínas de choque térmico (HSP) e antígenos específicos da espécie (12) [1].
A expansão das proteínas de choque térmico HSP 70 é um dos achados mais interessantes, com expansões independentes tendo acontecido em cada uma das espécies, resultando em 22 a 32 cópias completas em cada uma das espécie, o que é bem expressivo, especialmente, quando comparamos com as 6 cópias destes genes em moscas de fruta e apenas 2 em seres humanos. Expansões similares de genes HSP já haviam sido identificadas em ostras (HSP 110), associadas ao estresse de temperatura, e em células tumorais, associadas ao estresse proteotóxico. Estas alterações e outras características sugerem outros paralelos da infecção pelas larvas das tênias com o câncer, o que abre também novas perspectivas terapêuticas. Em ambos os caso ocorre proliferação descontrolada, invasão e metástase, sendo as células tumorais e os cistos difíceis de matar sem causar danos ao tecido circundante. Por isso, os pesquisadores esperam que os cisticercos possam ser vulneráveis a estratégias similares àquelas usadas no combate ao câncer; como a supressão da mitose, a indução da morte celular programa (apoptose) e a prevenção da replicação do DNA por parte das células do parasita/tumor. De fato, como os autores do artigo nos lembram, certos fármacos anti-helmínticos, como a niclosamida, mebendazol e albendazol já haviam sido demonstrados como capazes de inibir a proliferação de certos cânceres [1].
O outro artigo, publicado no dia 28 de abril, na revista Nature Genetics, nos fornece insights sobre a evolução das tartarugas (na verdade, 'Testudines', como tartarugas, jabutis, cágados, tigres d'água etc) e do seu plano corporal singular. O casco destes animais é realmente uma estrutura bastante particular, mesmo quando comparada a outros vertebrados encouraçados (como tatus e rinocerontes indianos), pois a carapaça das tartarugas é formada de vértebras e costelas modificadas, com suas escápulas exibindo uma topologia 'ao avesso', posicionadas contra a caixa torácica.
Além disso, estes animais não apresentam a fenestra do osso temporal em seus crânios, dificultando muito a reconstrução filogenética e o estabelecimento de sua posição relativa na árvore dos ´répteis' (sauropsídeos). Mesmo a utilização de dados moleculares não ajudou muito a avançarmos nesta questão e três hipóteses de relacionamento evolutivo ainda persistem [2, 3]:
As tartarugas seriam parentes bem próximos (um grupo irmão) do clado formado por lagartos, serpentes e pela Tuatara (Lepidosauria);
As tartarugas seriam mais aparentadas com Archosauria que inclui, como representantes modernos, os crocodilianos e as aves, as remanescentes dos dinossauros, e, por fim;
As tartarugas seriam um grupo de répteis bem antigo, que teria divergido muito cedo do resto do grupo, chamado de anapsida.
A partir dos rascunhos dos genomas de duas espécies de tartarugas, Pelodiscus sinensis (a tartaruga de caso mole) e Chelonia mydas (tartaruga marinha verde), os pesquisadores chegaram a conclusão da estreita relação das tartarugas com linhagem dos arcossauros, atualmente representadas pelas aves e crocodilos. Os dois grupos parecem que se separaram entre 267,9 e 248,3 milhões de anos atrás, entre o Permiano Superior e o Triássico [2].
Abaixo à direita, podemos ver a filogenia com base nos dados genômico das duas espécies de tartarugas (P. sinensis e C. mydas). Na filogenia são mostrados os tempos de divergência estimados de 12 espécies de vertebrados calculados a partir da primeira e segunda posição dos códons de 1113 genes que existem em cópias únicas e codificam proteínas presentes em todos estes grupos [MYA significa milhões de anos atrás]. A topologia da árvore, ou seja, o seu padrão de nós e ramos, é suportada por valores de bootstrap de 100% e por uma avaliação estatística suplementar não exibida aqui. As elipses pretas nos nós indicam os intervalos de confiança de 95% das distribuições posteriores estimada para os tempos de divergência [Para mais algumas informações sobre os métodos de inferência filogenética, vejam esta resposta de nosso tumblr]. Os círculos vermelhos indicam os tempos de calibração por meio de fósseis que são utilizados para definir os limites superior e inferior das estimativas [2].
Os cientistas também identificaram uma grande expansão dos genes de receptores olfativos e, mais curioso ainda, os resultados da expressão gênica durante a fase embrionária mostraram que há uma divergência em forma de ampulheta durante esta fase em comparação com a com a expressão gênica durante a fase equivalente na embriogênese de pintos. A conservação máxima nos padrões de expressão gênica, ou seja, correspondente ao ponto de afunilamento da ampulheta, ocorre por volta do período filotipico dos vertebrados, a chamada 'faringula', fase que sucede a gastrulação, ao invés de ocorrer em fases posteriores que mostram o padrão comum amniota [2]. [As figuras mosrando o esqueleto de uma tartaruga e as filogenias foram retiradas do artigo e do material suplementar de [Wang, Z et al. 2013]
Este resultados são importantes pois indicam quem apesar da morfologia bem particular das tartarugas, elas seguem o padrão de desenvolvimento básico embrionário dos vertebrados, passando pelo fase de faringula, e, só depois, desenvolvendo seu plano corporal sui generis, ao expressar na região dorsal do embrião genes também envolvidos na formação dos membros [2, 3].
Define-se o período filotípico como o ponto durante o desenvolvimento embrionário em que as características específicas do filo aparece no embrião que, tradicionalmente, é considerada a fase na qual os embriões de diferentes espécies pertencentes ao filo em questão mais se assemelham [Veja os artgos "Por que cinco dedos?" e "De determinantes 'genéricos' aos 'genéticos': A importância da física nos primódios da evolução animal."]. Nos vertebrados esta fase é conhecida como 'faringula' [Veja na figura abaixo e a esquerda, retirada de Bolker, 2001], a fase que se segue a formação da nêurula e na qual aparecem a maioria dos rudimentos de órgãos que ainda , entretanto, encontram-se pouco diferenciados. Entre as características principais desta fase, em vertebrados, temos a notocorda dorsal e o tubo neural, os somitos, as fendas branquiais na porção anterior do embrião e uma cauda na porção posterior. Muitas dessas características acabam desenvolvendo-se e mantendo-se nos adultos, mas outras não. Estas acabam regredindo ou dando origem a estruturas e órgãos distintos em diferentes grupos do filo. Esta fase, como já deve ter ficado claro, não ocorre no começo do desenvolvimento, mas mais adiante, representando um ponto de convergência a partir de inícios de embriogênese bem distintos, refletindo adaptações as estruturas presentes ou ausentes no ovo, como o saco vitelínico, ou aos diferentes mecanismos de gastrulação. Portanto, os embriões convergem para uma fase de maior similaridade relativa a partir de origens muito diferentes, e, após seus término, divergem novamente adquirindo as características específicas do grupo ao qual pertencem, o que gera uma forma de ampulheta nas trajetórias desenvolvimentais dos vertebrados [3].
Este período de gargalo da ampulheta seria produto das complexas interações locais, entre diferentes tecidos e regiões embrionárias que se constituiriam em módulos de desenvolvimento, que em fases mais precoces e tardias seriam mais autônomos, interagindo muito pouco uns com os outros nestas outras fases. Isso ocorreria desta maneira por que, como nesta fase é que surgem as características essenciais do filo, qualquer mudança ali poderia ter consequências graves e alterações nos padrões de expressão dos genes e dos processos de interação celular e tecidual que ocorrem nesta fase teriam repercussões mais globais e drásticas do que ocorreria caso alterações similares ocorressem em outros períodos [4].
Porém nas últimas décadas, o próprio conceito de uma fase filotípica em vertebrados passou a ser questionado, pelo menos, em suas formulações mais rigorosas. O biólogo do desenvolvimento Michael Richardson e vários colaboradores, mostraram que na realidade a faringula não é tão conservada assim entre os vertebrados. Por exemplo, os desenhos do naturalista alemão Ernst Haeckel dos embriões de vertebrados não refletem muito bem a realidade, pois eles são distorcidos para ajustarem-se as expectativas prévias de Haeckel, tanto com a retirada de elementos como sacos vitelínicos, que fariam os embriões parecerem menos similares, como com a modificação dos tamanhos e proporções dos embriões dos diversos vertebrados ilustrados, para torná-los mais parecidos uns com os outros do que realmente são [4].

Na realidade, parece não haver uma fase embrionária única, altamente conservada, em que todo o conjunto de características tipicas do filo surgem em todas as espécies estudadas. Embora, realmente os vertebrados passem por uma fase onde eles se parecem mais uns com os outros (apesar desta semelhança ser bem menor do que Haeckel acreditava) do que em outras fases, o tempo relativo de aparecimento de características tais como brotos de membros e primórdios órgãos varia de espécie para espécie. Estudos mais pormenorizados mostram que ao se aplicarem métricas mais objetivas, estas similaridades são ainda menores e não há fase onde não ocorram modificações nos tempo e ordem dos surgimentos das características e nem de suas posições relativas.
Mais recentemente, entretanto, outra virada ocorreu. Vários estudos sobre os padrões de expressão e a conservação dos genes expressões durante as fases filotípicas mostraram a consistência de um modelo de ampulheta, dando mais vigor ao ideia [4]. Acima à direita, temos uma figura retirada da versão online do livro de S.F. Gilbert [5] que por sua vez havia sido adaptada de um artigo de Richardson [Veja aqui]. Compare com os desenhos de Haeckel, logo à esquerda, e veja as diferenças que foram convenientemente deixadas de lado pelo naturalista alemão.
Porém, o modelo da ampulheta ainda não havia sido testado em outras espécies que não as dos organismos modelo, muito menos, um que apresenta características anatômicas tão peculiares como tartarugas. Neste novo estudo, os pesquisadores estenderam este teste ao desenvolvimento embrionário das tartaruga de casco mole comparando-o com o desenvolvimento de embriões de pintos, um organismo modelo bem conhecido e estudado. Eles, então, usaram os dados de RNA-seq e em um método de Bayes hierárquico, desenvolvido pelo próprio grupo em trabalho anterior, mostrando que os perfis de expressão gênica de todo embrião (GXPs) evidenciam, claramente, um padrão de ampulheta na embriogênese das tartaruga de casco mole e dos pintos. Valendo-se de várias medidas da divergência da expressão gênica entre os dois organismos em fases equivalente, os pesquisadores mostraram que por volta da fase TK11 da tartaruga e da fase HH16 dos pintos, há a maior conservação dos genes expressos, mesmo com a quantidade de expressão mantendo-se bem estável durante todo o desenvolvimento [2].
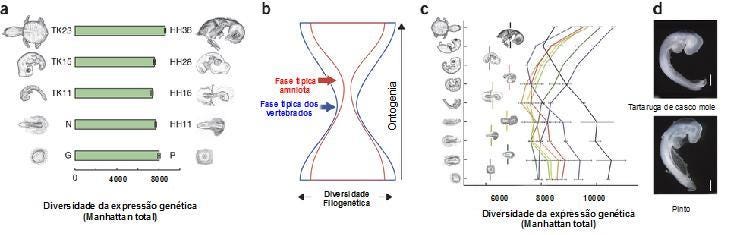
Na figura acima, em A, estão representadas as distâncias de GXPs de todo o embrião (médias aparadas dos valores M, TMM, controlados para a profundidade) de 11.602 genes ortólogos em estágios de desenvolvimento selecionados dos embriões de tartaruga de casco mole e do pinto [2]. As barras de erro, (desvio padrão, com o valor p = 7 × 10-7 sob heterocedasticidade) determinados pelo teste ANOVA. Em C pode ser visualizada uma comparação de tudo contra tudo das distâncias dos GXP dos embriões de tartarugas de casco mole (Manhattan total) que indica que a maior similaridade ocorre entre os embriões da tartaruga na fase TK11 e fase galinha HH16. Estes resultado, entretanto, não foi suficientemente robusto para confirmar que o estágio onde a conservação é máxima, ou seja, o ponto de estrangulamento da ampulheta, ocorre, realmente, na fase filotípica dos vertebrados em geral, aparentemente, podem ser um pouco mais deslocada para o fim da fase filotípica, o que ilustra a importância dos alertas de Richardson e outros pesquisadores e, ao mesmo tempo, sugere um padrão hierarquizados de modelos de ampulheta ao longo do desenvolvimento animal. A fase HH16, anteriormente, em outro estudo havia sido identificada com o período filotípico dos vertebrados, mas nesta comparação não coincide com o modelo apresentado no esquema do meio, B. Como podemos perceber, no modelo hipotético da 'ampulheta aninhada', tanto a divergência como uma recapitulação entre ontogenia e filogenia podem visualizadas.
A ideia é que, dependendo da proximidade das espécies comparadas, as fases mais conservadas do desenvolvimento podem ser diferentes. Assim as comparações entre os embriões vertebrados dão origem a fase filotípica (em azul), enquanto comparações dentro do grupo dos amniotas (em vermelho), um grupo mais restrito de vertebrados, dão origem a uma ampulheta com o ponto de estrangulamento ocorrendo posteriormente ao da fase filotípica. Em D vemos, de cima para baixo, um embrião de tartaruga de casco mole e do pinto, com as barras de escala de 1 mm. Outra descoberta importante é a expressão do gene Wnt, um gene envolvido com a formação de membros, na zona de crescimento do casco dorsal, o que dá apoio a ideia de que esta via tenha sido cooptação durante a evolução desta característica específica das tartarugas [2] Na figura abaixo [disponibilizada pelo Instituto Riken] podemos ver melhor algumas destas características.
De acordo com os cientistas, seus dados e análises sugerem que a evolução da tartaruga foi acompanhada por um período filotípico dos vertebrados inesperadamente conservador, seguido de uma repadronização, específica para a linhagem das tartarugas, do desenvolvimento embriológico para originar a nova estrutura do casco [2, 3].
Os avanços nas tecnologias e metodologias de sequenciamento de DNA de alta performance e a drástica redução dos custos destas técnicas têm permitido que acumulemos mais e mais informações sobre diferentes organismos a um ritmo atordoante. Porém, o sequenciamento genômico, por si só, não é um fim em si mesmo. As comparações entre genomas de espécies diferentes, a anotação de genes específicos (por meio da qual é possíve transferir a informação de genomas bem conhecidos para os novos), a descoberta de perdas e ganhos de genes e regiões genômicas (e outras modificações genéticas) em linhagens particulares, associadas com seus modos de vida e demografia, juntamente com estudos experimentais refinados da expressão gênica em diversas fases do desenvolvimento dos organismos, é que realmente nos permitem avançar na compreensão de como a evolução ocorre, além de poderem nos conduzir a descoberta de potenciais aplicações médicas e industriais destas informações.
Vivemos em uma época empolgante para as ciências da vida, especialmente para a biologia evolutiva, e devemos aproveitar estas oportunidades de compreender a história da vida, pois, afinal de contas, esta é, também, a nossa história.
----------------------------------------------------------
*O fenômeno do trans-splicing é um tipo diferente de processamento de transcritos de RNAs que ocorre em alguns eucariontes e que contrasta com a forma 'normal' de processamento chamada de (cis)splicing em que um RNA pré-mensageiro, isto é, transcrito diretamente a partir de uma sequência de DNA codificante eucarionte e contendo ainda os introns, tem estas porções de DNA não codificantes retiradas para dar origem ao transcrito maduro, o RNA mensageiro, que será, então, traduzido pelos ribossomos. No entanto, em alguns eucariontes, os mRNAs são formados pela união em conjunto de moléculas de transcritos RNA separadas,os genes adjacentes são transcritos como unidades continuas, uma unidade policistrônica (usado em oposição aos transcritos monocistrônicos que contem as sequencias codificadoras de apenas uma sequencia polipetídica), a semelhança do que ocorrem em bactérias. Porém, estes transcritos concatenados não podem ser traduzidos pelos ribossomos em proteínas, por isso, a necessidade de se separar estes transcritos em unidades monocistrônicas e prepará-los para a tradução através do trans-splicing, que é quando uma sequência líder curta idêntica, o tal spliced leader (SL), é emendada à extremidade 5' dos vários mRNAs, o que permite a tradução dos transcritos como unidades individuais, como os mRNAs tradicionais processados por meio do (cis)splicing.
Lodish H, Berk A, Zipursky SL, et al. Molecular Cell Biology. 4th edition. New York: W. H. Freeman; 2000.
Pagon RA, Bird TD, Dolan CR, et al., editors. GeneReviews™ [Internet]. Seattle (WA): University of Washington, Seattle; 1993-.
WormBook: The Online Review of C. elegans Biology [Internet]. Pasadena (CA): WormBook; 2005.
--------------------------------------------------
Referência:
Tsai IJ, Zarowiecki M, Holroyd N, Garciarrubio A, Sanchez-Flores A, Brooks KL, Tracey A, Bobes RJ, Fragoso G, Sciutto E, Aslett M, Beasley H, Bennett HM, Cai J, Camicia F, Clark R, Cucher M, De Silva N, Day TA, Deplazes P, Estrada K, Fernández C, Holland PW, Hou J, Hu S, Huckvale T, Hung SS, Kamenetzky L, Keane JA, Kiss F, Koziol U, Lambert O, Liu K, Luo X, Luo Y, Macchiaroli N, Nichol S, Paps J, Parkinson J, Pouchkina-Stantcheva N, Riddiford N, Rosenzvit M, Salinas G, Wasmuth JD, Zamanian M, Zheng Y, Taenia solium Genome Consortium, Cai X, Soberón X, Olson PD, Laclette JP, Brehm K, & Berriman M (2013). The genomes of four tapeworm species reveal adaptations to parasitism. Nature, 496 (7443), 57-63 PMID: 23485966
Wang Z, Pascual-Anaya J, Zadissa A, Li W, Niimura Y, Huang Z, Li C, White S, Xiong Z, Fang D, Wang B, Ming Y, Chen Y, Zheng Y, Kuraku S, Pignatelli M, Herrero J, Beal K, Nozawa M, Li Q, Wang J, Zhang H, Yu L, Shigenobu S, Wang J, Liu J, Flicek P, Searle S, Wang J, Kuratani S, Yin Y, Aken B, Zhang G, & Irie N (2013). The draft genomes of soft-shell turtle and green sea turtle yield insights into the development and evolution of the turtle-specific body plan. Nature genetics PMID: 23624526
Turtle genome analysis sheds light on turtle ancestry and shell evolution RIKEN Institute; sexta-feira, 26 de abril de 2013
Bolker, Jessica A (Apr 2001) Evolutionary Developmental Biology: Developmental and Genetic Mechanisms of Evolutionary Change. In: eLS. John Wiley & Sons Ltd, Chichester. doi: 10.1038/npg.els.0001061
Gilbert SF. Developmental Biology. 6th edition. Sunderland (MA): Sinauer Associates; 2000.
Créditos das Figuras:
SCIEPRO/SCIENCE PHOTO LIBRARY
EYE OF SCIENCE/SCIENCE PHOTO LIBRARY
