Ressuscitando genes e proteínas ancestrais: Uma viagem molecular no tempo.
Tudo o que sabemos sobre o passado mais remoto depende daquilo que restou dele nos dias atuais e do que podemos concluir a partir daí. As viagens no tempo da ficção científica, pelo menos como imaginadas por H.G. Wells e outros autores, ainda são só isso: ficção. Os paradoxos que se impõem a partir das indagações filosóficas sobre o tema e a gigantesca complexidade e as assustadoras demandas que as teorias da física moderna fariam, caso as viagens no tempo para o passado fossem possíveis, não nos deixam muita esperança, na prática, de voltar ao passado e testemunhá-lo por nós mesmos. Essa simples constatação tem por implicação que as ciências históricas (como a geologia, a paleontologia e boa parte da biologia evolutiva) deparam-se com limitações óbvias, que, não obstante, não nos impediram de alcançar conclusões muito sólidas sobre o que de fato ocorreu ou não.
Porém, apesar de as viagens no tempo para o passado continuarem apenas objeto da ficção científica, o avanço nas técnicas de sequenciamento, síntese, expressão e análise de biomolécula - juntamente com o desenvolvimento de métodos filogenéticos, equipamentos e procedimentos computacionais necessários a sua execução - estão nos dando a oportunidade de 'trazer do passado' biomoléculas há muito desaparecidas de nosso planeta. Essas técnicas têm modificado e enriquecido o estudo da evolução biológica. Elas nos permitem olhar muito mais longe em nosso passado evolutivo do que antes havíamos ser possível. Os genes e as proteínas de espécies extintas não estão mais para sempre perdidos no tempo.
Abordagens horizontais e as abordagens verticais:
Existem dois tipos principais de abordagens para o estudo da evolução molecular funcional. O primeiro tipo de abordagem envolve estudos comparativos e de mutagênese que concentram-se apenas nas versões atuais de proteínas semelhantes, de vários tipos de organismos aparentados, mas que exibem funções distintas. O segundo tipo de abordagem, como o nome sugere, envolve rastrearmos a história evolutiva das sequências de DNA e de proteínas ancestrais. Esse segundo tipo de abordagem vale-se das modernas técnicas de análise filogenética que permitem a criação de árvores de genes e a inferência dos estados ancestrais das sequências desses genes nos ancestrais comuns dos grupos de seres vivos que estão sendo estudados [1].
Essa segunda classe de estratégias permite que, não só identifiquemos as porções responsáveis pelas diferentes funções destes genes (e das proteínas por eles codificadas), mas também que desvendemos a ordem das mutações que deram origem às diferentes variantes, a partir das sequências ancestrais. Essas abordagens também nos abrem a possibilidade de estudarmos como mutações não diretamente ligadas a alteração de função dos genes podem ter influenciado, indiretamente, interferido nesse processo. Isso quer dizer que a reconstrução de sequências ancestrais possibilita o estudo dos efeitos epistáticos entre diversas mutações ao longo de uma sequência de aminoácidos de uma proteína. Através delas podemos investigar como essas mudanças permitiram que as atuais funções dessas biomoléculas evoluíssem a partir de funções ancestrais [1].
Mas temos que ir com calma. Primeiro de tudo, precisamos compreender melhor porque os estudos horizontais são extremamente limitados.
Olhando apenas para o presente:
"A razão porque estudos deste tipo fracassam é que eles ignoram a história. A função da proteína evoluiu a medida que mutações acumularam-se ao longo do tempo, verticalmente - em linhagens ancestrais de proteínas, enquanto que as comparações horizontais de proteínas modernas envolvem apenas as pontas da árvore evolutiva.” [1]
Dois são os problemas principais com a abordagem horizontal. Para começar, muitas diferenças nas sequências de aminoácidos entre duas proteínas modernas (da mesma família, mas com funções distintas) são irrelevantes para as diferenças funcionais, tendo acumulado-se durante o período em que as funções de interesse simplesmente não se alteraram. A deriva genética aleatória - especialmente em populações pequenas - pode levar a fixação e, portanto, ao acúmulo de mutações neutras e até mesmo de algumas ligeiramente desvantajosas. Porém, mesmo que essas mutações não alterem a função corrente da biomolécula, elas, ainda assim, podem alterar o contexto bioquímico e biofísico da qual a função depende. Esse é o outro ponto muito importante. Modificações que não alterem as funções das sequências de DNA e de seus produtos (ou que as alterem muito pouco) são invisíveis a seleção natural purificadora, mesmo que elas provoquem mudanças na forma como a função é alcançada - por exemplo, a partir de pequenas alterações nas interações intramoleculares entre os resíduos de aminoácidos [2]. Essas mudanças podem, ainda assim, tornar mudanças posteriores (provocadas por mutações adicionais) possíveis, ou, em contrapartida, inviabilizá-las completamente [1, 2].
Mutações restritivas e permissivas:
Duas variedades de mutações epistáticas ao longo de caminhos evolutivos são particularmente relevantes. As chamadas 'mutações permissivas' introduzem aminoácidos necessários para que uma proteína tolere outras mutações, essas sim, fundamentais para mudança de função. Essas mutações podem, por exemplo, aumentar a estabilidade da proteína, 'tamponando-a' contra a aquisição (por mutações subsequentes) de novos resíduos de aminoácidos desestabilizadores, mas que criariam uma função distinta [1, 2].
O outro lado da moeda são as 'mutações restritivas'. Esse tipo de mutações introduz resíduos de aminoácidos que são incompatíveis com as funções de outros membros da família, isto é, uma vez na sequência de aminoácidos elas impedem que sequência desempenhe outra função observada em uma outra proteína bem similar, de alguma linhagem próxima, mesmo que ela sofra uma alteração idêntica a da outra proteína. Normalmente, isso acontece porque tais mutações produzem 'confrontos estéricos', isso é, as cadeias laterais dos aminoácidos trocados podem obstruir as cadeias laterais de outros resíduos e, desta maneira, interferir com o enovelamento da cadeia de aminoácidos, impedindo que a proteína adquira uma conformação tridimensional final estável e consistente com essa outra função* [2]. Por isso, caso apenas troquemos os resíduos que acreditamos serem os responsáveis pelas diferenças de função (como aqueles que encontram-se 'sítios de ligação' ou dos 'centros ativos', de receptores e enzimas) de uma outra proteína (em cuja linhagem mutações restritivas ocorreram ou em que mutações permissivas não ocorreram), isso poderá resultar em uma proteína não funcional ou cuja função é bastante prejudicada, mesmo que os resíduos trocados tenham sim um papel essencial na mudança funcional [1] e representassem nas proteínas ancestrais biomoléculas viáveis. Isso mostra que o pano de fundo mutacional é muito importante para eventuais mudanças de função.
Em resumo, isso quer dizer que, as versões atuais das proteínas (isto é, aquelas existentes nos organismos remanescentes) podem simplesmente não serem capazes de trocar de funções umas com as outras sem que essas outras mudanças no pano de fundo (que ocorreram em versões ancestrais intermediárias destas sequências) também ocorram, o que só pode ser descoberto pelos métodos verticais [1, 2]. Toda essa rica história de interações indiretas é perdida nas abordagens horizontais.
Por causa disso, estudos levando em conta apenas as biomoléculas modernas - que permitem investigarmos o efeito de mutações apenas no pano de fundo das sequências atuais - podem facilmente perder este efeito da história mutacional das sequências (em diferentes ramos de uma linhagem). Efeito esse que teria permitido cada variante evoluir suas funções específicas [1].
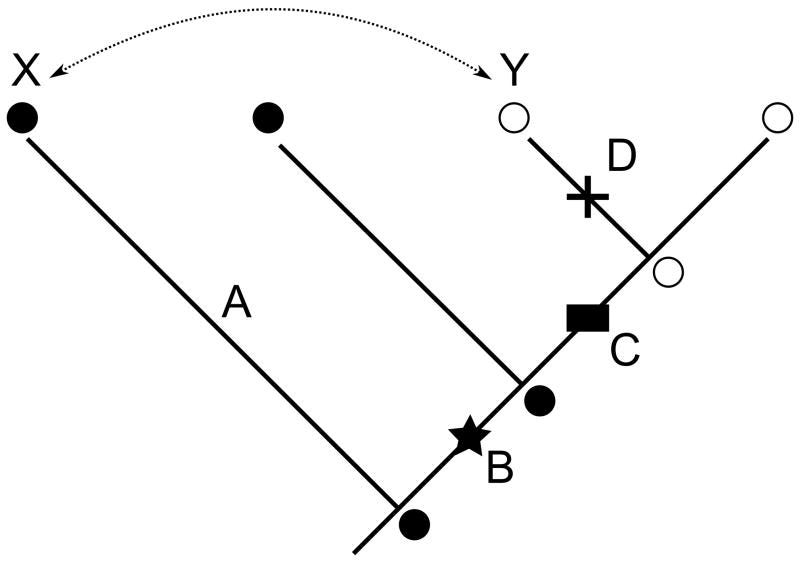
Na figura acima (retirada de 1) podemos visualizar a 'dissecação' dos fatores que determinam as funções de uma sequência dentro de uma família de proteínas que exibem uma função ancestral (representada pelo círculo preenchido) e uma função derivada (representada pelo círculo branco). Veja que a alteração levou à função derivada (nova) foi causada por um subconjunto das alterações na sequência ao longo do ramo C (retângulo preto). No cenário mostrado aqui, foram necessárias mutações permissivas no ramo B (estrela preta). Elas permitiram que a proteína tolerasse as mutações que levaram a mudança de função. Veja que mutações restritivas - incompatíveis com a função ancestral - acumulam-se no ramo D (cruz). É aí é que encontra-se o principal problema das abordagens horizontais. Nestas abordagens, tudo que ocorre é simplesmente a colocação de resíduos de aminoácidos que são responsáveis pela função de uma proteína moderna em outra proteína moderna, que tem uma função diferente. Isso é, exatamente, o que está representado pela seta. É por isso que tal abordagem é tremendamente ineficiente. A nova função não surgiu dessa maneira. Tal perspectiva ignora a história evolutiva. As duas sequências atuais diferem em todas as mutações ao longo de A, B, C e D. Porém, o mais importante é que a proteína X não tem as mutações permissivas e, portanto, não pode passar a desempenhar a função derivada. Por fim, a proteína Y, ao longo de sua evolução, adquiriu mutações restritivas, que, assim, não permitem que ela tolere a função ancestral, o que inviabiliza a mudança de Y para X [1].
É por isso que, como explicam Harms e Thornton [1], uma abordagem na qual explicitamente leve-se em conta a filogenia do grupo de organismos cujas biomoléculas estão sendo investigadas é essencial. Uma estratégia vertical desse tipo iria concentrar-se naquelas mutações que ocorreram ao longo do ramo na árvore genealógica em que ocorreu a alteração funcional de interesse. Essa abordagem é claramente mais eficiente porque somente as mutações que ocorreram durante um período limitado de tempo evolutivo precisam ser investigadas. Ao utilizarmos apenas o pano de fundo mutacional da linhagem da proteína na qual a sequência de alterações realmente ocorreu, evita-se a confusão causada por causa de interações epistáticas. Inclusive isso permite identificar as eventuais mutações epistáticas restritivas e permissivas envolvidas no processo.
Reconstrução de sequências ancestrais:
A grosso modo, o que se faz é inferir a sequência de aminoácidos da proteína ancestral por meio de métodos filogenéticos e a partir daí, utilizando-se o conhecimento do código genético, cria-se uma sequência de DNA correspondente, sintetizasse-a e a insere em algum tipo de célula, de algum sistema de cultura celular in vitro, para que seja transcrita em um mRNA e traduzida em uma proteína. Uma vez expressa, a proteína ancestral pode ser testada diretamente, usando-se ensaios bioquímicos e farmacológicos, ou purificada, de modo que possa ser analisada por métodos como a difração de raio X ou ressonância nuclear magnética (NMR), além de métodos de bioinformática estrutural, que permitem criar um modelo da estrutura tridimensional da proteína. É possível fazer isso com todos os intermediários, que podem, então, serem testados e comparados entes si e com as versões atuais exibidas pelos representantes remanescentes das linhagens sendo investigadas [1, 3].
Ao lado, vemos resumida a estratégia de 'ressurreição de genes ancestrais'. No esquema podem ser vistas as etapas necessárias para 'ressuscitar' e caracterizar um gene ancestral e seu produto primário. É mostrada na figura uma proteína hipotética de algum vertebrado ancestral, como um exemplo [3].
As sequências disponíveis para as várias versões da proteína de diferentes espécies evolutivamente aparentadas são alinhadas juntamente com as sequências de um 'grupo externo', que é como são chamadas na sistemática filogenética as sequências que são consideradas mais distantemente relacionadas as sequências empregadas na análise (as sequências do 'grupo interno') do que estas últimas são umas das outras. A partir daí - e utilizando-se de métodos específicos - uma árvore filogenética do grupo é construída. Caso já haja uma filogenia robusta do grupo em questão, as sequências das diversas espécies são simplesmente sobrepostas a árvore filogenética disponível [1, 3, 4, 5].
Uma vez de posse da filogenia do grupo, são empregados métodos filogenéticos, como máxima parcimônia, máxima verossimilhança ou bayesianos [3, 4]. Esses métodos são utilizados para inferir a melhor estimativa do estado ancestral para cada sítio (posição) na sequência, tendo como base os dados das sequências atuais. Os métodos de máxima parcimônia operam minimizando a quantidade de mudanças evolutivas ao longo dos ramos da árvore filogenética, com as árvores (ou estados ancestrais) que requerem o menor número de alterações sendo os preferidos [Para saber mais sobre os métodos cladísticos veja ’Filogenia Mastigada 1: Princípios de Filogenia e conceitos básicos’, ’Filogenia Mastigada 2: Polarização de Séries de Transformações e o conceito de Homoplasia’, ’Filogenia Mastigada 3. Grupos Monofiléticos e Merofiléticos e a filosofia por detrás da Filogenia’, ’Filogenia Mastigada 4 : Interpretando uma árvore filogenética – parte ½’ e ’Filogenia Mastigada 5 – Interpretando uma árvore filogenética 2/2’].
O problema principal com esses métodos é que eles não incorporam um modelo evolutivo explícito, mesmo que certas versões, como os métodos de parcimônia ponderada, possam acomodar diferentes cenários de mudança de caráter. Esse método também não é apropriado quando ocorreram múltiplas substituições em um sítio, já que assume implicitamente uma baixa taxa de mudança [4, 5]. Já os métodos filogenéticos baseados na análise de máxima verossimilhança utilizam como critério de ‘otimalidade’ um escore de verossimilhança, que é calculado tendo como base um modelo de evolução molecular especifico. O escore de otimização pode ser usado para especificar a topologia [padrão de ramificação] e os outros parâmetros da árvore, como os comprimentos de ramos, as frequências dos estados das características e os próprios estados ancestrais. Por fim, os métodos bayesianos também podem ser utilizados para calcular os estados ancestrais por meio das probabilidades posteriores. Tais probabilidades podem ser calculadas usando-se as topologias, os comprimentos de ramificação e os parâmetros de modelo, todos estimadas por máxima verossimilhança, que são inseridos como probabilidades prévias (‘priors’) ou, de maneira alternativa, as probabilidades posteriores podem ser calculadas considerando a incerteza na topologia e nos parâmetros obtidos por máxima verossimilhança, empregando o que os especialistas chamam de 'Métodos de Monte Carlo via Cadeias de Markov (MCMC)'. A vantagem de tais métodos é que, ao utilizarem um modelo explícito de evolução molecular, isso nos permite incorporar o conhecimento dos mecanismos e restrições que atuam sobre as sequências codificantes, além de podermos comparar o desempenho de diferentes modelos, tornando a análise muito mais realista e robusta [3, 4, 5]. Esses métodos estocásticos permitem que os pesquisadores explorarem diferentes modelos de evolução molecular de maneira que eles consigam determinar o quão robustos são os resultados da reconstrução ancestral, Isso é extremamente importante, uma vez que modelos simplificados ou irrealistas podem, em certos casos, produzir reconstruções filogenéticas enganosas ou incorretas, o que deixa claro a importância de métodos e critérios rigorosos para a seleção de modelos. Para saber mais sobre essas questões dê uma olhada nesta resposta aqui, disponível em nosso tumblr.
Esses métodos são então usados para inferir a melhor estimativa do estado ancestral para cada sítio (posição) na sequência tendo como base os dados das sequências atuais. Uma fez feito isso, são sintetizadas pequenas sequências de DNA (chamadas de 'oligonucleotídeos') que são montadas em genes que codificam a proteína ancestral através da técnica de PCR gradual ou por digestão de restrição/ligação. Nesta etapa pode ser utilizada a estratégia de mutagênese dirigida ao local caso a sequência ancestral possa ser criada através da introdução de apenas algumas alterações num gene existente. Essas sequências de DNA são então inseridas em vetores especiais e introduzidas em culturas de células por métodos de 'transformação' o que permite que elas sejam expressas e estudadas [3, 4].
De acordo com Thornton [3], a preferência é sempre dada as sequências de aminoácidos porque elas contêm menos "ruído" do que as sequências de DNA, que são mais sujeitas a convergência e a reversão mutacional. Com base em nosso conhecimento do código genético (a relação entre tripletos de nucleotídeos, os códons, e os aminoácidos especificados por eles nas proteínas) é inferida uma sequência de DNA codificante para a proteína ancestral. Caso o sistema de expressão específico possua algum tipo de preferência de códons (viés de códons), isso ser pode ser introduzido para melhorar a taxa de tradução da proteína, de maneira, que ela seja obtida em maior quantidade. A proteína pode depois disso ter suas funções caracterizadas por meio de testes experimentais, bioquímicos (tais como ensaios 'gene repórter', que permitem que estudemos sua expressão) ou farmacológicos (como os 'ensaios de ligação', em é quantificada a ligação da proteína ao substrato ou ao ligante). Em outros casos pode-se investigar a resposta da proteína a algum parâmetro físico, como a luz, ou sua estabilidade termodinâmica [3, 4, 5]. Como já mencionado, caso necessário, também é possível purificar a proteína ancestral para melhor estudar sua estrutura tridimensional. Isso é feito através da clonagem do gene ancestral em um plasmídeo, que permite a expressão de alto nível, que é transfectado para células bacterianas ou de mamíferos em cultura.
Rodopsinas Ancestrais e a visão dos dinossauros:
Com o intuito de compreender melhor a evolução da visão entre os Arcossauros, Belinda Chang e alguns outros cientistas resolveram examinar a proteína responsável pela visão em baixas condições de iluminação [4, 5]. Os arcossauros são um grupo de vertebrados que inclui os dinossauros já extintos, bem como as aves (o grupo remanescente de dinossauros terópodes), além dos crocodilianos modernos e ancestrais [4, 5]. Os ancestrais desses animais, que formam um dos principais ramos dos répteis diapsidas, originaram-se por volta de 240 milhões de anos atrás, no começo do período Triássico.
Chang e seus colaboradores [4, 5] usaram como base de sua análise as sequências de quatro espécies de animais, pertencentes a três grupos de arcossauros remanescentes: Crocodilianos (Aligátor); Aves (Pombo e Galinha), Peixe (Paulistinha), além das sequências de 26 outros vertebrados que foram utilizados como grupos externos [Veja aqui para maiores detalhes] [3]. A equipe liderada por Chang usou três tipos diferentes de modelos de máxima verossimilhança (modelos baseados em nucleotídeos, baseados em aminoácidos e em códons). As proteínas atuais diferiram em no máximo de 16% em relação as suas sequências de aminoácidos, o que permitiu que a sequência proteica do arcossauro ancestral fosse reconstruída com pouca ambiguidade, utilizando os três tipos de dados [3]. Para determinar qual modelo melhor se ajusta aos dados, os pesquisadores sempre que foi possível fizeram testes de razão de verossimilhança dentro de cada tipo de modelo [4, 5].
Feita a análise inicial, os pesquisadores constataram que as reconstruções do nódulo ancestral dos arcossauros, usando os modelos mais adequados de cada tipo, estavam todas de acordo, com exceção de três resíduos de aminoácidos, em relação aos quais uma reconstrução diferia das outras duas. Para determinar se estes pigmentos ancestrais seriam funcionalmente ativos, os genes correspondentes foram quimicamente sintetizados e depois expressos numa linhagem de células de mamífero em culturas de tecido. Isso foi feito através da montagem de sequência de DNA de 1 kilobase, que codifica a proteína rodopsina ancestral (a partir de cinco oligonucleotídeos longos), que foi, então, clonada em um vetor de expressão sob o controlo de uma sequência promotora constitutiva. O vetor foi transfectado em uma cultura de células de mamífero. Após a expressão deste gene, garantida pelo promotor constitutivo, e a subsequente tradução dos mRNAs resultantes, em um proteína, nas células em cultura, essas biomoléculas foram purificadas e submetidas a ensaios funcionais in vitro; os mesmo normalmente empregados para caracterizar as rodopsinas comuns [3].
Os produtos desses genes artificiais ligavam-se a molécula 11-cis-retinal, produzindo pigmentos fotoativos estáveis com o pico de absorção (λmax) de luz por volta da faixa dos 508 nanômetros, ligeiramente desviada para a porção vermelha do espectro em relação ao que se observa nos pigmentos dos vertebrados existentes atualmente. Os pigmentos do arcossauro ancestral também ativaram a transdução da proteína G [veja aqui para compreender como funciona a proteína G] retiniana, medida num ensaio de fluorescência [4, 5]. E isso ocorreu a uma taxa semelhante ao que acontece com a rodopsina bovina [3]. Isso tudo indica que foi obtida uma molécula completamente funcional.

Ao lado [figura retirada de 4], em A podemos ver uma linha do tempo dos períodos geológicos, onde são mostradas as idades aproximadas de amostras antigas usadas em estudos que tentam amplificar material genético antigo, com as estimativas da idade do gene ancestral reconstruído da rodopsina do Arcossauro ancestral também indicada. Em B podemos observar uma filogenia das rodopsinas dos vertebrados utilizados para a reconstrução do nódulo ancestral, que está também indicado na figura. A topologia da árvore reflete a compreensão atual das relações sistemáticas entre as principais linhagens de vertebrados, com os comprimentos de cada ramo e os parâmetros dos modelos estimados através de máxima verossimilhança sendo indicados. Os picos de absorção aproximados das diversas rodopsinas dos vertebrados estão ressaltados em itálico.
Tais resultados são consistentes com a hipótese de que o arcossauro ancestral possuía a habilidade - pelo menos ao nível molecular - de ver bem em luz fraca. Isso significa que ele poderia ter sido sido ativo durante à noite. Esse resultado por si só é impressionante. Agora dispomos de janela para ecologia destas criaturas extintas.Podemos investigar alguns detalhes de seus modos de vida que nunca poderiam ter sido vislumbrados a partir do simples exame dos fósseis ou de qualquer outra evidência não molecular sobre o comportamento de dinossauros e de outros arcossauros extintos [3].
A ressurreição de receptores hormonais ancestrais:
Joe Thornton, a partir de 2003, seguiu uma abordagem semelhante. Desde então, ele e seus colaboradores vêm ampliando e aprofundando seus estudos. Seu trabalho iniciou-se com a análise das sequências de genes para receptores de hormônios esteroides de vários animais vivos hoje em dia. Essas sequências foram alinhadas e usadas para 'descer' a filogenia deste grupo de animais, usando os métodos explicados anteriormente. A partir dai, a equipe de Thornton pode inferir a sequência mais provável do antepassado comum de todos esses receptores, que deve ter existido entre 600 e 800 milhões de anos atrás. A reconstrução dos estados de tais sequências e a síntese das proteínas correspondentes (que permitiu ‘ressuscitar' tais biomoléculas) possibilitou sua equipe "testasse experimentalmente hipóteses sobre a evolução que de outra forma seria apenas especulação" [6]. Através destes estudos, eles puderam mostrar que o receptor ancestral era sensível aos estrogênios, mas não a outros hormônios relacionados - apoiando a ideia de que esta família de receptores teria evoluído por meio de vários eventos de duplicações de genes, com as cópias evoluindo gradualmente suas afinidades por outros tipo de moléculas ligantes [6].
Thornton e seu grupo de pesquisa decidiram investigar em maior profundidade dois receptores para esteroides estreitamente relacionados, o receptor mineralocorticoide (MR), que liga ao hormônio aldosterona e que temo um papel na regulação de eletrólitos e água; e o receptor para glicocorticoides (GR), ao qual liga-se o cortisol e que está relacionado ao controle da resposta ao estresse. As evidências indicam que estas duas moléculas surgiram a partir da duplicação de genes que aconteceu há mais de 450 milhões de anos. Porém, a aldosterona só surgiria muitos milhões de anos depois, resultando em um enigma [6]:
'Como a seleção poderia conduzir a evolução de ‘uma fechadura’ (o MR) para encaixar em uma chave (a aldosterona) que ainda não existia?' [6]
A resposta para essa pergunta foi descoberta pela equipe de Thornton através de um trabalho liderado pela pesquisadora Jamie T. Bridgham [6, 7]. O fato de o antepassado de ambos os receptores ser sensível à aldosterona sugeria que existia algum outro ligante mais antigo com uma estrutura similar. Desta maneira, quando a aldosterona surgiu, esta molécula pode ser recrutada durante a evolução pelo receptor já existente (que por ligar-se a um molécula similar, estava 'pré-adaptado') para exercer uma nova função biológica. Esse processo, Thornton chamou de ‘exploração molecular’ [6, 7]. Nesse mesmo trabalho, os cientistas também mostraram que o receptor para glicocorticoides estava evoluindo suas próprias funções específicas [6, 7].
Mais pistas sobre a evolução desses receptores continuaram ser descobertas em trabalhos subsequentes, revelando mais detalhes sobre como a evolução opera ao nível molecular. A equipe de Thornton explorou a história de como o GR tornou-se sensível ao cortisol em um processo que demorou cerca de 20 milhões de anos, de acordo com suas estimativas. Para isso eles, trabalhando em colaboração com biólogos estruturais, que determinaram a estrutura cristalina do ancestral comum do GR e do MR. O que eles descobriram foi que apenas duas mutações cruciais em conjunto são capazes de alterar o sítio de ligação do receptor ancestral de modo a fazê-lo ligar-se preferencialmente ao cortisol. Além dessas duas, outras cinco mutações foram necessárias para produzir as versões atuais e garantir a especificidade aos ligantes atuais. Porém, quando os pesquisadores tentaram reverter a evolução das sequências, revertendo as sete mutações, elas não conseguiram transformá-lo de volta em uma proteína funcional, como era a versão ancestral. [Lembra-se de toda aquela discussão sobre mutações restritivas e permissivas?].
A nova molécula era quase completamente incapaz de responder a qualquer hormônio, ilustrando a limitação das abordagens horizontais que discutimos lá em cima, no começo dessa postagem. Foi quando eles perceberam que, além dessas sete mutações, um punhado de outras mutações também surgiram durante esse processo e, mesmo não estando diretamente associadas a afinidade e especificidade ao ligante, foram necessárias para produzir o receptor específico de cortisol. Thornton mostrou que era necessário desfazer essas mutações também para reverter a mudança [6]. De acordo com Thornton, tais mutações agiram como uma catraca evolutiva, impedindo que o receptor recuperasse sua função ancestral. Essa é uma demonstração fantástica de como o caminho da evolução pode ser historicamente contingente e depender de vários eventos completamente aleatórios [6, 7]. Nas palavras do próprio Thornton:
"O acaso desempenha um papel muito grande na determinação de quais resultados evolutivos são possíveis", diz ele [6].
"A evolução abre portas para o futuro. Mas parece fechá-los - firmemente - por trás dele também", disse um editorial do New York Times [citado em 6].
Esses resultados sugerem que o receptor de esteroides ancestral tinha um perfil de especificidade semelhante aos dos receptores de estrogênio modernos; com os ligantes hormonais dos outros receptores (e as afinidades pelos genes-alvo com quem eles ligam-se e aos quais regulam) emergindo mais tarde, como novidades evolutivas derivadas. Porém, a conclusão mais surpreendentemente talvez seja a que, como o primeiro receptor na família era ativado por um hormônio que aparece apenas ao final de uma via de síntese de esteroides (que exibe vários intermediários, como a progesterona e testosterona), os novos pares 'hormônio-receptor' foram evoluindo conforme as sequências dos genes de receptores ancestrais foram sofrendo duplicação [6, 7]. Desta maneira, apenas posteriormente, evoluíram maior afinidade por esses esteroides que já estavam presentes, mas que, inicialmente, eram apenas passos intermediários da via de síntese, e só mais tarde passariam a desempenhar novas funções [3]. Essas descobertas ilustram a natureza oportunista e contingente do processo evolutivo. Logo após as duplicações dos genes que codificavam esse receptores, os receptores extras devem ter permanecido órfãos ou redundantes. Porém, devido as similaridades estruturais entre os ligantes originais e seus diversos intermediários nas suas vias de síntese, eventualmente, mutações que levassem a ligação preferencial entre um desses intermediários e a cópia mutante do receptor - induzindo sua ativação em alguma nova circunstância (ao mesmo tempo que uma das cópias mantinha a função original) que trouxesse quaisquer vantagens - permitiriam o começo da evolução de uma nova função [7]. Em seu artigo de 2006 [7], os pesquisadores explicam que a ligação entre a aldosterona e o MR evoluiu de maneira gradual, completamente consistente com a moderna teoria evolutiva. O que mudou foram as funções que estavam sendo selecionadas ao longo do tempo. Como já ressaltado a sensibilidade do AncCR à aldosterona estava presente desde o inicio, muito antes do hormônio ter surgido. Ela era portanto um mero subproduto de restrições específicas no receptor associadas a ativação pelo seu ligante original, que tinha semelhanças com a aldosterona.
Como também vimos, após a duplicação que deu origem ao GR e ao MR, apenas duas substituições na linhagem que deu origem ao GR foram necessárias para produzir receptores com diferentes perfis de resposta hormonal. A evolução posterior do MR, que poderia ser regulado independentemente pela aldosterona, permitiu uma resposta endócrina mais específica do controle da homeostase de eletrólitos, mas sem também desencadear a resposta ao estresse associada ao GR; e, de modo complementar, as respostas de estresse não interfeririam com a homeostasia hídrica e de sais [6]. Veja que este cenário evolutivo – no qual um antigo receptor recruta um novo ligante - é o oposto da dinâmica previamente estabelecida para os receptores androgênicos e de progesterona (AR, PR), onde as duplicatas de um antigo receptor, que respondia a estrogênio, evoluíram afinidade a esteroides que anteriormente serviam como intermediários na síntese do próprio ligante ancestral [7].

Acima podemos observar um esquema da evolução da especificidade da sinalização MR-aldosterona por meio da ‘exploração molecular’. Em (A) está mostrada a via de síntese de hormônios corticosteroides. Nela as moléculas ligantes para o receptor corticoesteroide (CR) ancestral e para os MRs existentes estão sublinhadas. O cortisol, que é o ligante para o GR dos tetrápodes está mostrando com uma linha logo acima de seu nome e a adição terminal da aldosterona está ressaltada com a cor verde. Os asteriscos mostram as etapas catalisadas pela enzima citocromo P-450 11b-hidroxilase. Note que apenas a enzima dos tetrapodes pode catalisar o passo marcado com um asterisco verde, dado a mutações específicas que ocorreram nessa linhagem. Em (B) podemos ver que a sensibilidade do MR à aldosterona precedeu o surgimento do próprio hormônio, já que o ancestral vertebrado não sintetizava aldosterona (círculo pontilhado), mas produzia outros corticosteroides (círculo cheio). Esses animais possuíam um único receptor com afinidade para ambas as classes de ligantes. A duplicação genética (em azul) deu origem a GR e MR e duas alterações na sequência do GR (vermelho) aboliram sua capacidade de ser ativado pela aldosterona, mas mantiveram sua sensibilidade ao cortisol [ver (C)]. Nos tetrápodes, a síntese de aldosterona teve origem devido à modificação da enzima citocromo P-450 11b-hidroxilase. Em (C) podemos inferir a base mecanicista para perda de sensibilidade à aldosterona nos GRs. Os gráficos mostram o que ocorre quando são introduzidas alterações de aminoácidos (que filogeneticamente servem de diagnostico que ocorreram durante a evolução de GR) no domínio de ligação do ligante do receptor ancestral (AncCR-LBD) por mutagênese. As curvas dose-resposta são mostradas para aldosterona (verde), para DOC (azul), e para cortisol (vermelho). Veja que o mutante duplo (lado inferior direito) tem um fenótipo semelhante ao visto no GR moderno. As setas mostram caminhos evolutivos através de um intermediário não funcional (vermelho) ou funcional (verde), Ali fica claro como a ordem das mutações foi importante para produzir a transição [6].
Em conjunto, essas duas histórias sugerem uma dinâmica evolutiva bem mais geral em que novas interações surgem quando uma molécula recém gerada – que pode ser uma ligeira modificação estrutural ou duplicada de uma previamente existente - recruta como um ligante (parceiro de interação) uma molécula mais antiga, que havia sido previamente restringida pela seleção para uma função inteiramente diferente [7].
“O enigma que os sistemas complexos representam para a evolução darwiniana depende da premissa de que cada parte não tem função - e, portanto, não pode ser selecionada para - até que todo o sistema esteja presente. Este quebra-cabeça poderia realmente fazer que a teoria de Darwin desmoronasse caso as funções das partes devessem permanecer estáticas todo o tempo. Mas praticamente todas as moléculas podem participar e participam em mais de um processo ou interação, então os elementos de um complexo podem ter sido selecionados no passado para funções não relacionadas. Nosso trabalho indica que os sistemas firmemente integrados podem ser montados combinando moléculas antigas com papéis ancestrais diferentes, juntamente com outras novas - geradas pela duplicação de genes ou elaboração de caminhos enzimáticos - que representam ligeiras variantes estruturais de elementos mais antigos. Propomos que a exploração molecular seja predominante tema na evolução, que pode fornecer uma explicação geral de como as interações moleculares críticas para a complexidade da vida surgiram a moda darwiniana.” [7]
Os trabalhos das equipes de Thornton, Chang e de outros biólogos evolutivos têm nos permitido compreender cada vez melhor como a evolução de sistemas biomoleculares complexos acontece. Além das novas técnicas e métodos que permitem trazermos o passado 'de volta', esses cientistas mostram como as objeções criacionistas esvaem-se frente a pesquisa científica moderna, deixando claro que tais objeções baseiam-se em formas muito estreitas e até caricatas de conceber a evolução biológica e que, para serem mantidas, dependem de ignorar a literatura científica e algumas das pesquisas mais impressionantes que tem sido realizadas nas últimas décadas.
--------------------
* Essas mudanças das relações epistáticas - isto é, do padrão de interferência entre diferentes resíduos de aminoácidos ao longo de uma mesma sequência proteica - podem ocorrer não só pela fixação de mutações neutras ou ligeiramente deletérias, em virtude da deriva genética, mas como resultado da interação da deriva com a seleção natural. Como mutações ligeiramente desvantajosas podem ser fixadas, reduzindo a aptidão do fenótipo associado àquela biomolécula, isso favorece a fixação por seleção natural de eventuais mutações compensatórias que aumentam a aptidão conferida pela sequência, podendo, porém, alterar as relações epistáticas entre os diversos resíduos [2]. A interação entre pressão de mutação, deriva genética em virtude de variações dos tamanhos das populações, seleção natural purificadora, seleção natural variante e por mutações compensatórias mostra que as vias de evolução de sistemas biomoleculares dependem muito de fatores estocásticos.
---------------------
Imagem da rodopsina de autoria de B. C. Chang disponível aqui.
--------------------
Referências:
Harms MJ, Thornton JW. Analyzing protein structure and function using ancestral gene reconstruction. Current opinion in structural biology. 2010;20(3):360-366. doi:10.1016/j.sbi.2010.03.005.
Ohta, Tomoko (February 2013) Molecular Evolution: Nearly Neutral Theory. In: eLS. John Wiley & Sons, Ltd: Chichester. doi: 10.1002/9780470015902.a0001801.pub4
Thornton JW. Resurrecting ancient genes: experimental analysis of extinct molecules. Nat Rev Genet. 2004 May;5(5):366-75. :10.1038/nrg1324
Chang, B.S.W. et al. Recreating a functional ancestral archosaur visual pigment. Molecular Biology and Evolution, 2002 Sep;19(9):1483-9. PubMed PMID: 12200476.
Chang, B.S.W. Ancestral Gene Reconstruction and Synthesis of Ancient Rhodopsins in the Laboratory Integr. Comp. Biol. 2003, 43 (4): 500-507 doi:10.1093/icb/43.4.500
Pearson H. Prehistoric proteins: Raising the dead. Nature. 2012 Mar 21;483(7390):390-3. doi: 10.1038/483390a.
Bridgham JT, Carroll SM, Thornton JW. Evolution of hormone-receptor complexity by molecular exploitation. Science. 2006 Apr 7;312(5770):97-101. DOI: 10.1126/science.1123348
