Semelhanças e diferenças entre os genomas e transcriptomas dos animais.
Os seres humanos guardam semelhanças impressionantes com outros animais tão distintos como as moscas de fruta e os vermes nematodes, com os quais compartilhamos um ancestral comum que viveu há mais de 540 milhões de anos. Contudo, além das semelhanças já bem conhecidas entre os números de genes, as sequências dos genes, e das proteínas que eles codificam (e mesmo do padrão de organização espacial de certas famílias multigênicas), novos estudos revelam outras similaridades, desta vez no próprio parão de expressão de muitos destes genes e nas redes regulatórias que eles estão envolvidos, normalmente associados com as regiões do DNA onde ligam-se os fatores de transcrição e elementos que controlam o estado da cromatina [(Ilustração de Darryl Leja, NHGRI, NIH].
As sequências de DNA associadas aos genes precisam ser expressas na forma de RNAs funcionais (como os microRNAs regulatórios), ou, como é mais conhecido, na forma de RNAs mensageiros (que serão traduzidos na forma de peptídeos e proteínas. Estas biomoléculas, por sua vez, combinam-se para fazer várias estruturas biológicas, agem no metabolismo modulando reações químicas ou interagem entre si e como outras pequenas moléculas não proteicas formando redes de sinalização intra e intercelular que controlam a morfologia das células e dos tecidos que elas formam, modulam a composição da matriz ao seu redor, além de controlarem as taxas de proliferação e morte celular, suas propriedades mecânicas, seu movimento e, assim, seus padrões de migração. Este processo se dá de modo bem organizado, com certos conjuntos de genes formando redes de interação metabólica ou de sinalização, através de seus produtos. Estas redes (ou 'módulos') não apenas regulam certas atividades bioquímicas e celulares mais gerais, como controlam também a expressão de seus próprios (e outros conjuntos de) genes. Isso é feito, por exemplo, por meio de expressão de proteínas chamadas de fatores de transcrição que ligam-se às regiões não codificantes do DNA, os elementos cis-regulatórios (como promotores, reforçadores, repressores etc) que, quando associadas aos fatores de transcrição, modulam a transcrição gênica, ligando-a e desligando-a, ou mesmo, aumentando ou diminuindo sua intensidade em diferentes situações e contextos. Estes conjuntos de genes e redes regulatórias são essenciais no processo de desenvolvimento embriológico, estando, por exemplo, por trás da formação dos padrões corporais gerais de cada tipo de organismo [Para saber mais sobre isso veja "É a evolução genética previsível? Parte I" e "É a evolução genética previsível? Parte II ou Além da genética parte I"].
Uma gigantesca análise de dados de expressão genômica liderada por pesquisadores da universidade de Yale e outros estudos conduzidos por cientistas das Universidades de Harvard e Stanford, todos recentemente publicados na revista Nature, mostram claramente que vermes e moscas, apesar das diferenças óbvias, conservam aquilo que muitos biólogos chamam de 'kits de ferramentas moleculares': conjuntos de genes co-expressos (e suas vias bioquímicas e de sinalização correspondentes) que são empregados durante o seu desenvolvimento ontogenético e portanto na formação dos seus padrões corporais característicos. Os pesquisadores puderam inclusive utilizar os padrões de expressão para alinhar as fases de desenvolvimento dos nematodes e das moscas, conseguindo encontrar uma nova correspondência entre uma fase do embrião do verme e a pupa da mosca, além daquelas correspondências embrião-embrião e larva-larva que já eram conhecidas.
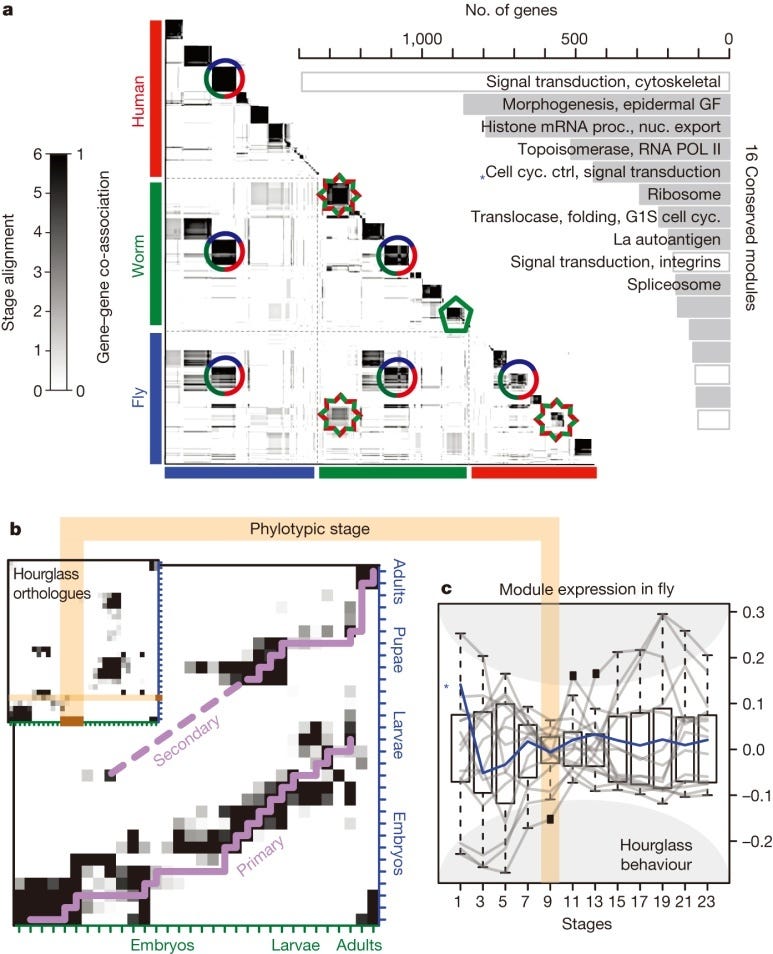
Na figura ao lado vemos à esquerda uma matriz de associação gene-gene de seres humanos, vermes nematodes e mosca-de-fruta na qual a coloração mais escura reflete o aumento da probabilidade (verossimilhança) de que um par de genes sejam atribuídos a um mesmo módulo. Um bloco escuro ao longo da diagonal representa um grupo de genes dentro de uma espécie. Caso isso esteja associado a um bloco fora da diagonal, então é um módulo inter-espécies (por exemplo, um módulo de três espécies conservadas é mostrado como um círculo e um módulo de verme-mosca é mostrado por uma estrela). No entanto, se um bloco da diagonal não tem associações fora da diagonal então ele forma um módulo espécie-específico de (por exemplo, pentágono verde). A direita é mostrado o enriquecimento funcional da Ontologia de Genes para os genes conservados dentro dos 16 módulos. [GF, fator de crescimento; nuc, nuclear.; proc., processamento. Em b, observamos os alinhamentos primários e secundários dos estágios de desenvolvimento verme-mosca realizados com base em todos os genes ortólogos (com a mesma origem ancestral) entre vermes e moscas]. Em seguida temos o alinhamento verme-mosca dos estágios usando apenas ortólogos da fase da ampulheta que é mais significativo e apresenta um 'gap' (marrom) correspondente ao estágio chamado fase filotípica [Veja para maiores detalhes Genomas e mais genomas em abril de 2013: Parasitas e Tartarugas e Por que cinco dedos?]. A escala do 'mapa de calor' em b é indicada no lado esquerdo, em um alinhamento de estágio destacado. Em c, vemos a expressão normalizada dos módulos conservados na mosca que mostra a menor divergência intra-organismo durante a fase filotípica (castanho). Um módulo representativo é indicado com um asterisco azul em a e c. [Nature 512, 445–448 (28 August 2014) doi:10.1038/nature1].
De fato, em um dos artigos os seus autores também relataram que, nos três organismos estudados, é possível prever os níveis de expressão gênica (tanto de elementos codificantes como não-codificantes) por meio de um "modelo universal", construído a partir de características da cromatina nas regiões promotoras (aquelas nos quais ligam-se proteínas, como fatores de transcrição e a RNA polimerase, que controlam a expressão dos genes), que utiliza apenas um único conjunto de parâmetros, independente do organismo. Porém, um outro estudo feito pelo mesmo pessoal de Yale, publicado em separado em artigo da revista PNAS, documenta diferenças genéticas extensas entre as várias regiões genômicas destas várias linhagens, sendo bastante particulares da história evolutiva de cada uma delas. Os autores, por exemplo, relataram que o complemento de pseudogenes do genoma humano reflete uma explosão maciça de atividade retrotransposons ('genes móveis' que usam intermediários de RNA para se proliferarem pelo genoma) que teria ocorrido na aurora da evolução dos primatas, diferentemente do que ocorre com os pseudogenes do verme e da mosca, que são reflexo de uma história de cópias extras inativadas. Porém, nos três casos, os pesquisadores puderam observar que pseudogenes mantinham um nível consistente de expressão, por volta de 15% deles sendo transcritos.
Estes resultados não são um paradoxo, mas ilustram um princípio básico da evolução molecular. Enquanto regiões com alta relevância funcional e estrutural tendem a ser mantidas por meio da seleção natural purificadora, as modificações nestas regiões normalmente ocorrem de maneira conservadora e indireta, por meio, principalmente, da duplicação de genes funcionais e da divergência mutacional das sequências das cópias que se dá através de processos como a subfuncionalização, neofuncionalização, originação gênica e por efeitos de dose. No entanto, não é geralmente isso que acontece com a maioria das sequências que perdem sua relevância funcional e estrutural. Em geral, elas transformam-se em pseudogenes, verdadeiros fósseis moleculares. A 'pseudogenização' é o resultado mais provável da duplicação de um gene, uma vez que a maioria das cópias extras é deteriorada e inativada por mutações. Eventualmente, um gene, mesmo sem ser duplicado, que era antes funcional também pode tornar-se um pseudogene caso ele perca da sua utilidade, por exemplo, por uma mudança ambiental que passa a prover o recurso que antes era fornecido apenas por um processo de conversão bioquímica, guiado pelo gene em questão. Porém, o mais importante é que quando ocorre a pseudogenização, diferentemente das sequências gênica e regulatórias funcionais, há uma tendência à divergência, com as sequências passando a refletir muito mais a história contingente particular de cada linhagem, após a separação de outras linhagens, com sua evolução passando a ser muito mais governada por processos estocásticos, como a deriva genética e o efeito carona.
"Por um lado, vimos semelhanças que refletem a necessidade biológica e, por outro lado, as diferenças que espelhavam a história do organismo", disse Cristina Sisu, do grupo de Yale e primeira autora do artigo do PNAS.
Estes resultados mostram como grandes diferenças nos padrões corporais podem ser obtidas a partir dos mesmos genes (e dos módulos gênicos básicos que eles constituem), que foram duplicados e reformulados de maneira contingente ao longo da evolução de cada grupo, mas, mesmo assim, mantendo certas semelhanças cruciais. Estas semelhanças, por sua vez, são um reflexo tanto de uma origem comum, como provavelmente também de 'princípios biológica de construção' universais, muito deles associados a restrições na forma como novas funções e estruturas surgem, ou seja, a partir de outras pré-existentes e, portanto, da organização genético-desenvolvimental prévia. Ao mesmo tempo, o estudo dos pseudogenes deixa claro que aquelas regiões que não estão diretamente envolvidas com a organização e funcionamento mais básico dos organismos estão bem mais livres para divergir e acumular as marcas da história e do acaso.
--------------------------------------
Referências:
Hathaway, Bill Evolution used similar molecular toolkits to shape flies, worms, and humans Yale News, August 27, 2014
Gerstein, Mark B. et al Comparative analysis of the transcriptome across distant species. Nature, 2014; 512 (7515): 445 DOI: 10.1038/nature13424
Araya, Carlos L. et al Regulatory analysis of the C. elegans genome with spatiotemporal resolution Nature, 2014 512, 400–405 28 DOI: 10.1038/nature13497
Brown, James B. Diversity and dynamics of the Drosophila tanscriptome Nature 2014 512, 393–399 doi:10.1038/nature12962
Ho, Joshua W. K. et al Comparative analysis of metazoan chromatin organization Nature, 2014 512, 449–452 doi:10.1038/nature13415
Boyle, Alan P. et al. Comparative analysis of regulatory information and circuits across distant species Nature, 2014 512, 453–456 doi:10.1038/nature13668
Sisu, Cristina et al. Comparative analysis of pseudogenes across three phyla PNAS 2014 [published ahead of print August 25, 2014] doi:10.1073/pnas.1407293111
